
Introducción
Mi nombre es Alexander Sadovnikov , soy un graduado del programa de maestría corporativa de ITMO y JetBrains en Desarrollo de Software y también un desarrollador senior de software bioinformático en el Departamento de Biología Computacional de BIOCAD .
— . : .
, , , , . - . , , . , . , , . , , , , .
, , , , .
, . , , . , , . , , . , !
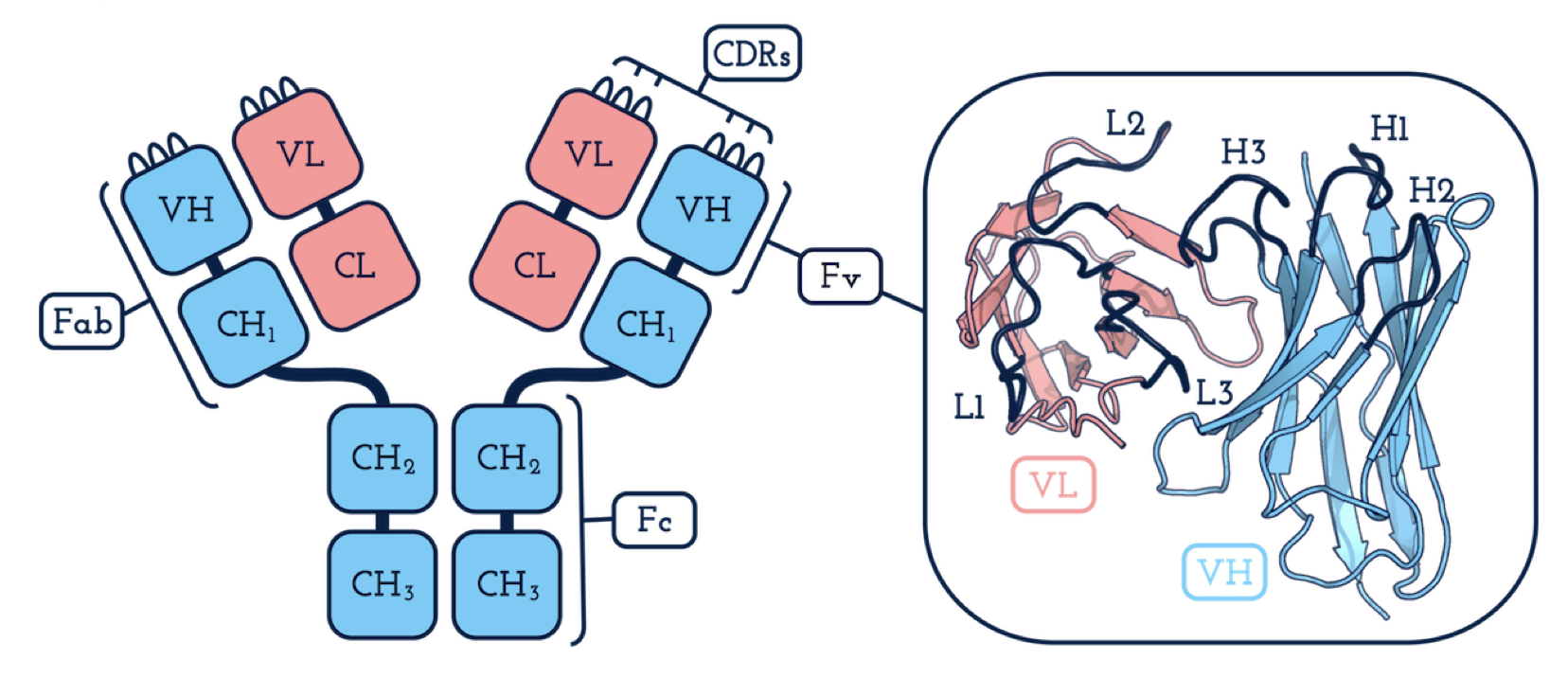
. ( L1, L2, L3 H1, H2, H3). : — -! . .
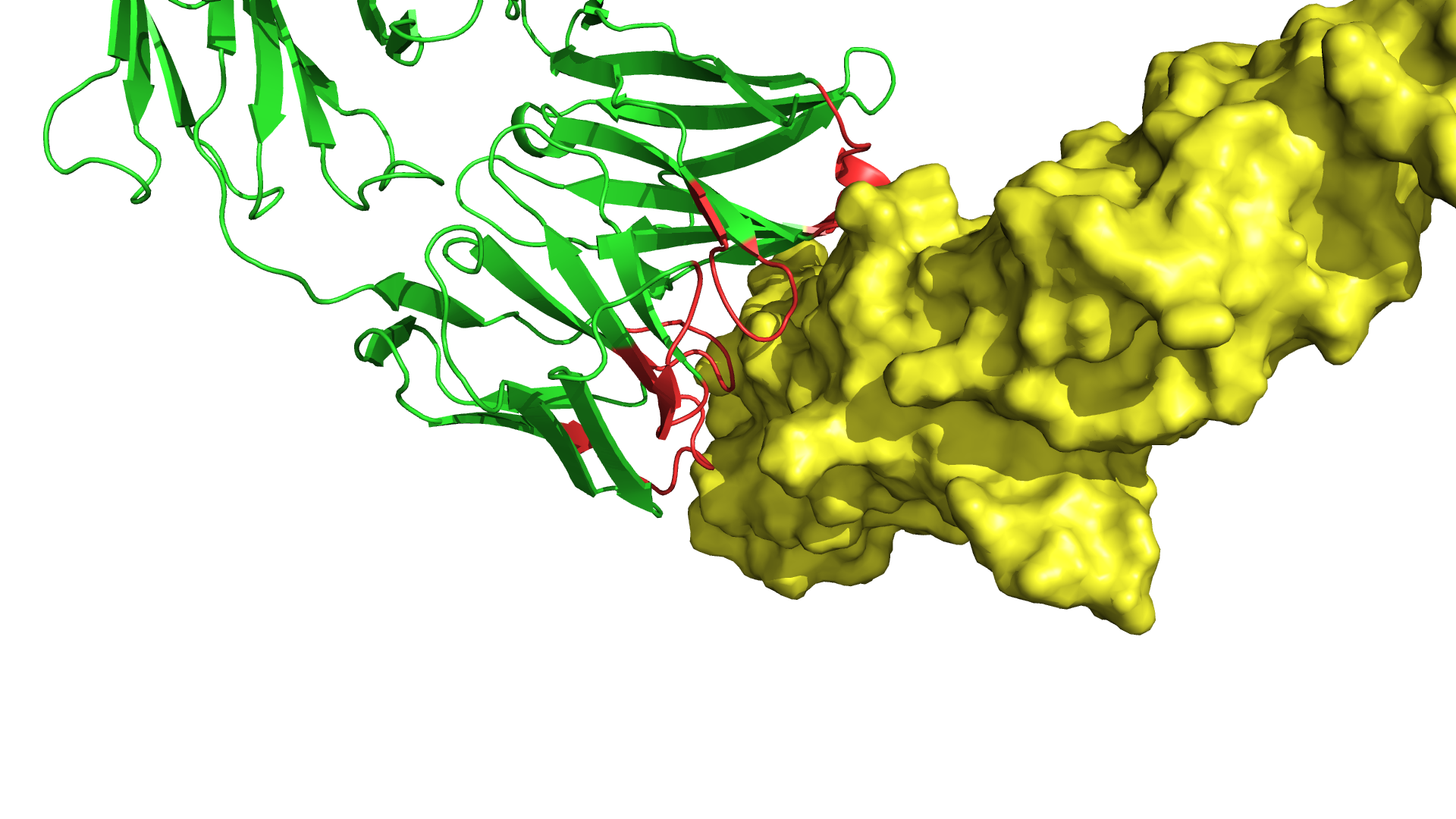
. , — . .
, . , , . , , , — , . , . , .
, . , , — , . , , . . , , . , .
, , . , , . , , , , Piper, .
. , . , . , , .
, , : , . , , . .
. . , . , .
, :
complexes = []
#
for rotation in rotations:
#
for shift in shifts:
#
#
moved_mol2 = rotate_and_shift(mol2, rotation, shift)
# ,
#
new_complex = (mol1, moved_mol2)
complexes.append(new_complex)
#
complexes.sort(lambda candidate_complex: score_complex(candidate_complex)) . , , , . — . , R — , S — . . .
1992 , , . .
, , , . :
1 , , . , 0 1, , , . 0 . .
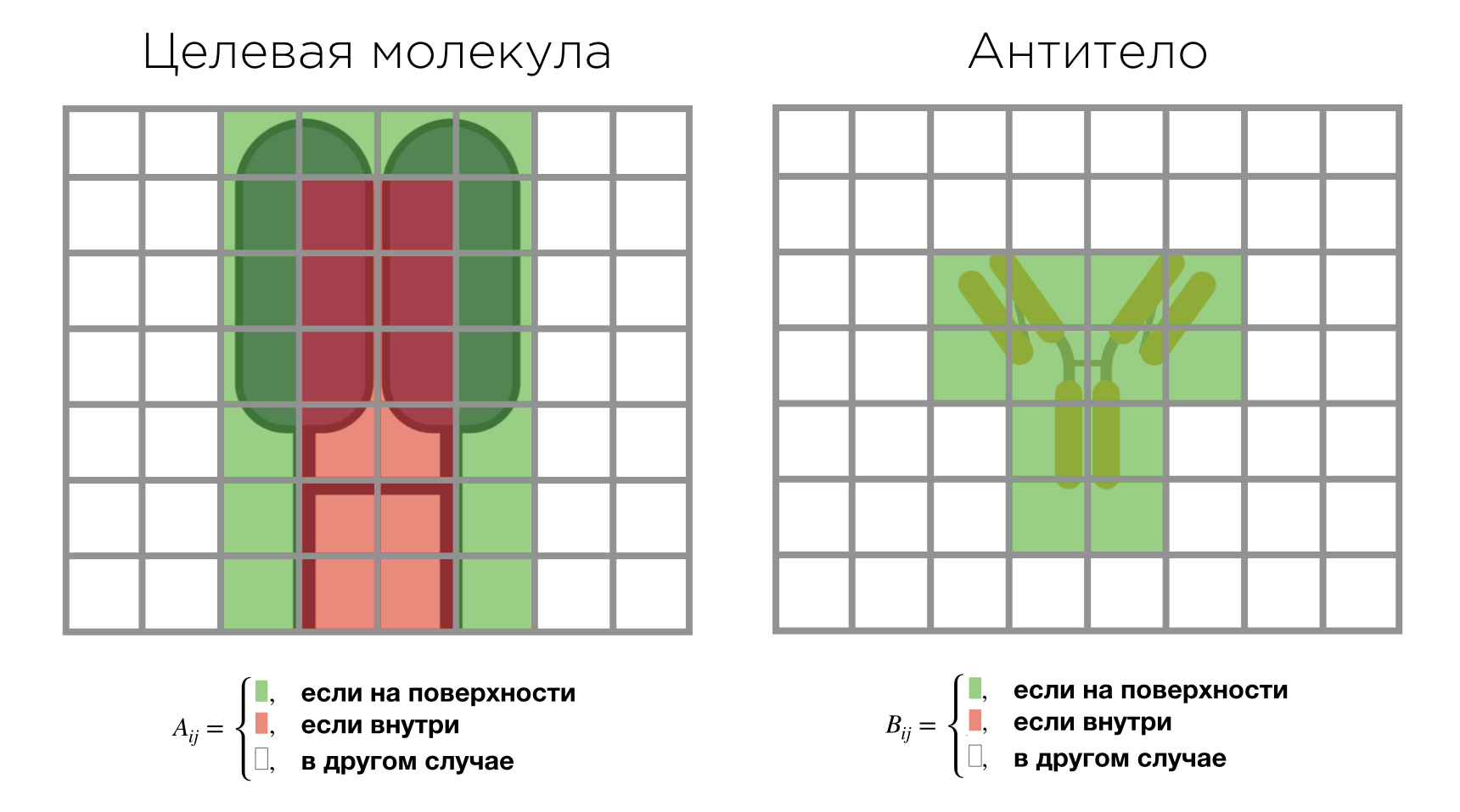
. , . , , . , 1. , , , . , , .
, . , , , , .
: , N — , . . , . , . . : . , , N, .
- . ( ), . , FFT. , , .
- :
complexes = []
#
grid_for_mol1 = build_grid(mol1)
#
fft_mol1 = fft_3d(grid_for_mol1)
#
for rotation in rotations:
#
rotated_mol2 = rotate(mol2, rotation)
#
grid_for_mol2 = build_grid(rotated_mol2)
#
fft_mol2 = fft_3d(rotated_mol2)
# .
#
# . , ,
#
correlations = ifft_3d(fft_mol1 * fft_mol2)
# ,
#
rotation_complexes = map(lambda shift_: (mol1, shift(rotated_mol2, shift_)))
#
#
complexes += list(zip(rotation_complexes, correlations))
#
complexes.sort(lambda (candidate_complex, score): score) , . , , , .
, , , . , , .
. , , , , .
, Piper, . — , , . :
— , — .
. , Piper : DARS (Decoys as Reference State) aADARS (asymmetric Antibody DARS). aADARS , DARS — .
, . aADARS, . , . — WAASP (Widely Applicable Antibody Statistical Potential). WAASP HEDGE, , :
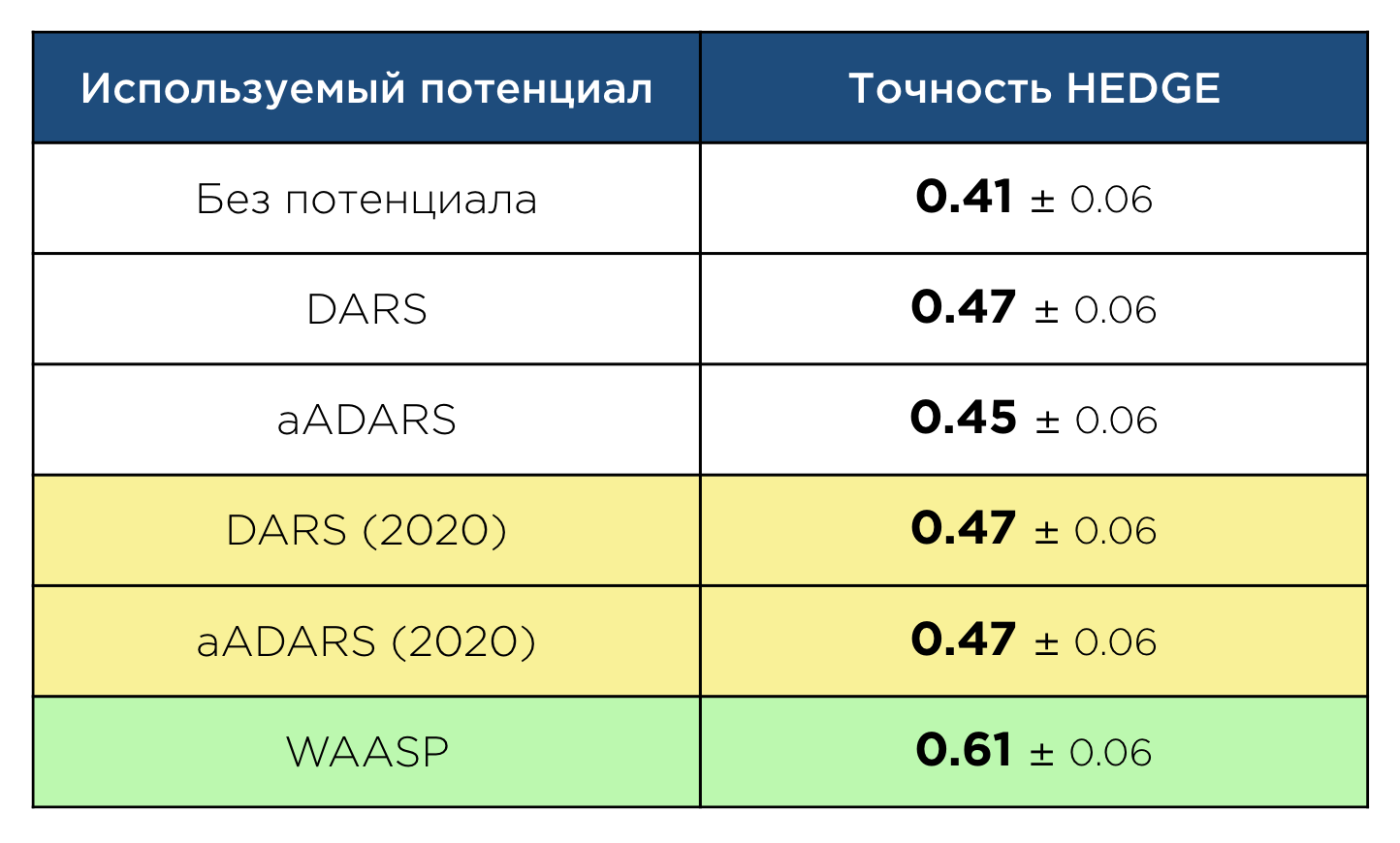
HEDGE,
, , , , DARS aADARS.
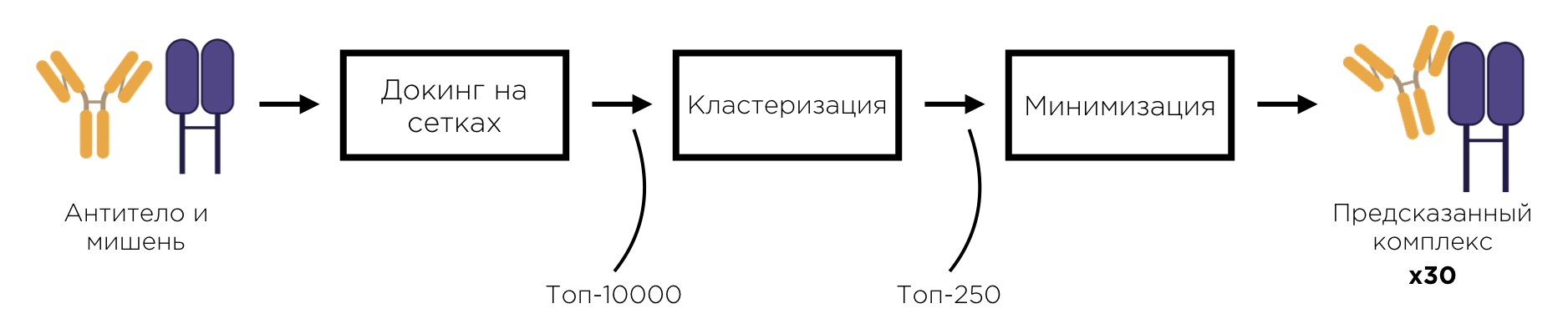
, . , , , . -10000 , , . 10000 , .
. , : , , . , . , , . , .
. . . RMSD (Root-Mean-Square Deviation). RMSD .
, , , , . , : , , — . , . , . , .
250 , . — , . , 10000 250, . .
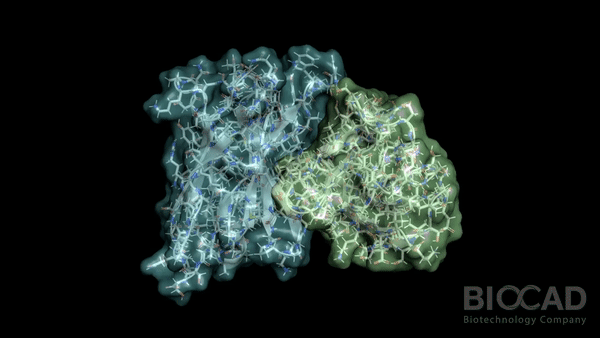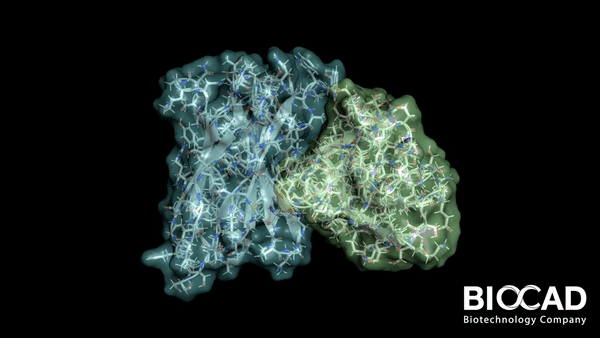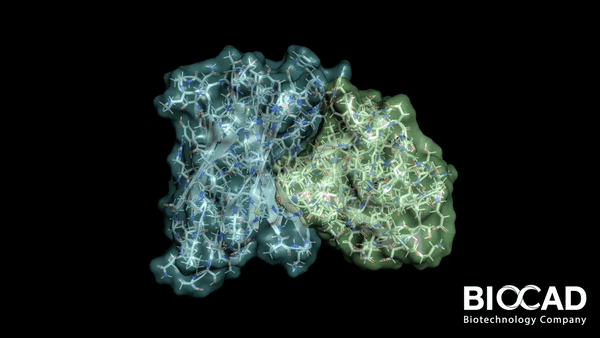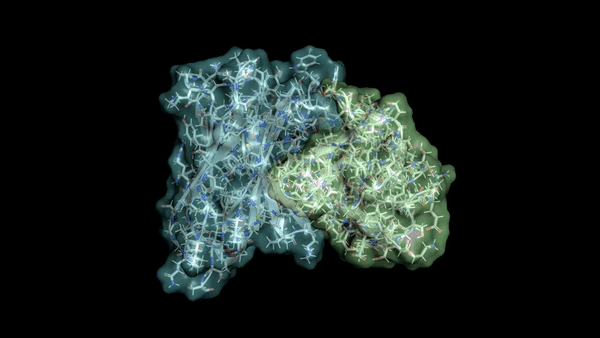
. , !
, . , , . . , , . , , , (, , !). , .
, , , , , . , , : — -, , ! , - , , .
, , : . , .
-, - , . -, , , , , .
, . . , , , .
, . , . , -, , , , .
. !
: , , , , — ! . , (, , ), : , .
. , — . , . , .
, , : . . , : , , , . , .
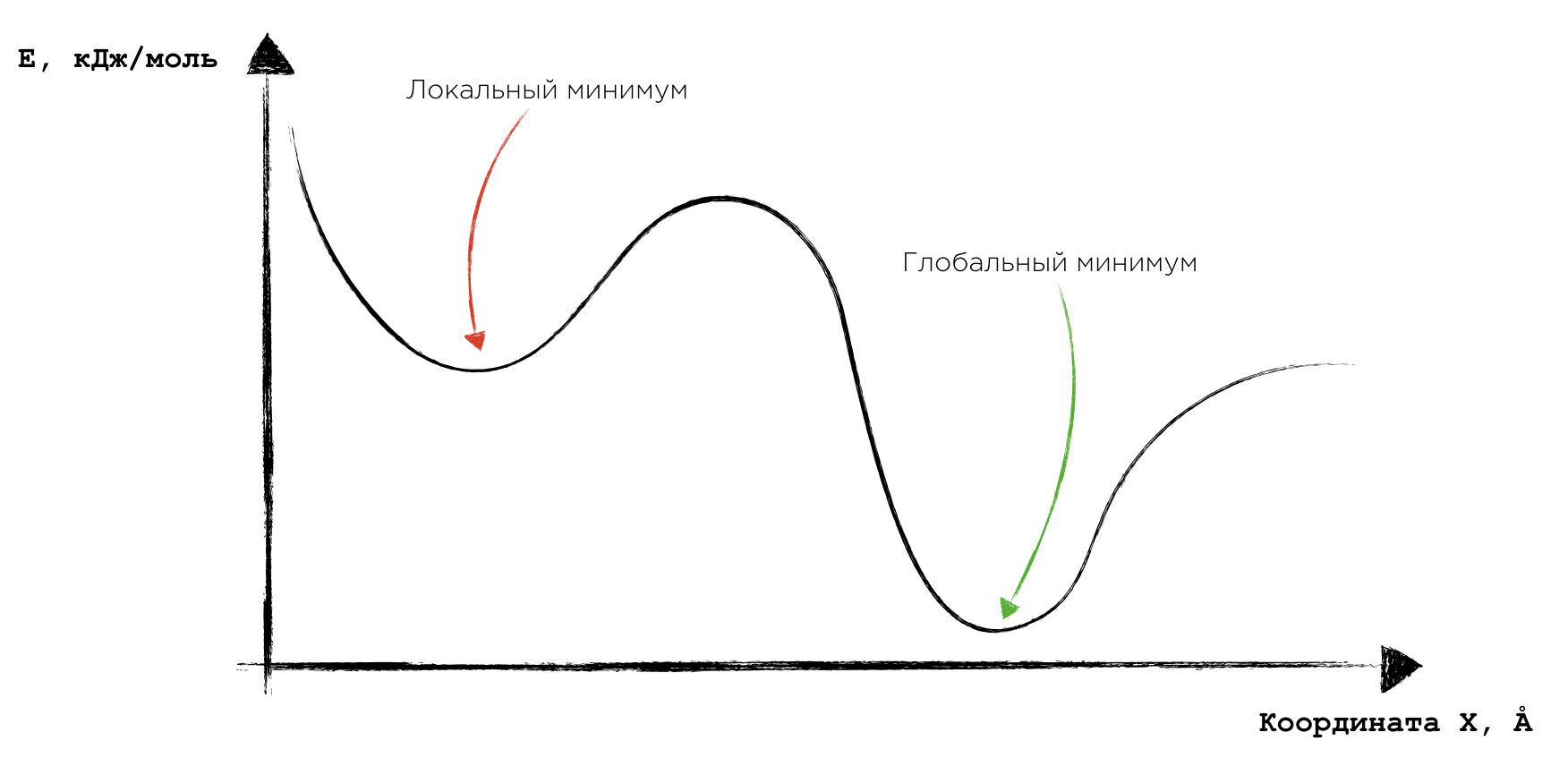
,
, . , . : - , ( ). , , . , .
. , , , . 30 , .
, !
, .
, . . , . : - !
, , , ( , ) . , , — . , , ( !) .
, , . , , . . . , , — .