Una vez, en la víspera de la conferencia de clientes, que se realiza anualmente por el grupo DAN, estábamos pensando en qué cosas interesantes podemos pensar para que nuestros socios y clientes tengan impresiones agradables y recuerdos del evento. Decidimos hacer un archivo de miles de fotos de esta conferencia y varias pasadas (y había 18 en total para ese momento): una persona nos envía su foto, y en un par de segundos le enviamos una selección de fotos con él durante varios años desde nuestros archivos.
No inventamos una bicicleta, llevamos la conocida biblioteca dlib a todos y recibimos incrustaciones (representaciones vectoriales) de cada persona.
Agregamos un bot de Telegram por conveniencia, y todo fue genial. Desde el punto de vista de los algoritmos de reconocimiento facial, todo funcionó de maravilla, pero la conferencia terminó y no quería separarme de las tecnologías probadas. Queríamos pasar de varios miles de personas a cientos de millones, pero no teníamos una tarea comercial específica. Después de un tiempo, nuestros colegas tenían una tarea que requería trabajar con cantidades tan grandes de datos.
La pregunta era escribir un sistema inteligente de monitoreo de bots dentro de la red de Instagram. Aquí nuestro pensamiento dio lugar a un enfoque simple y complejo:
forma simple: consideramos todas las cuentas que tienen muchas más suscripciones que suscriptores, sin avatar, el nombre completo no está completo, etc. Como resultado, obtenemos una multitud incomprensible de cuentas medio muertas.
El camino difícil: dado que los robots modernos se han vuelto mucho más inteligentes, y ahora publican publicaciones, duermen e incluso escriben contenido, surge la pregunta: ¿cómo atraparlos? En primer lugar, vigile de cerca a sus amigos, ya que a menudo también son bots, y rastree fotos duplicadas. En segundo lugar, rara vez un bot sabe cómo generar sus propias imágenes (aunque esto es posible), lo que significa que las fotos duplicadas de personas en diferentes cuentas en Instagram son un buen disparador para encontrar una red de bots.
¿Que sigue?
Si un camino simple es bastante predecible y rápidamente da algunos resultados, entonces un camino difícil es difícil precisamente porque para implementarlo tendremos que vectorizar e indexar volúmenes increíblemente grandes de fotografías para comparaciones posteriores de similitudes: millones e incluso miles de millones. ¿Cómo ponerlo en práctica? Después de todo, surgen preguntas técnicas:
- Velocidad de búsqueda y precisión
- Espacio en disco ocupado por los datos
- El tamaño de la RAM utilizada.
Si solo hubiera unas pocas fotos, al menos no más de diez mil, podríamos limitarnos a soluciones simples con agrupación de vectores, pero para trabajar con grandes volúmenes de vectores y encontrar los vecinos más cercanos a un determinado vector, se requieren algoritmos complejos y optimizados.
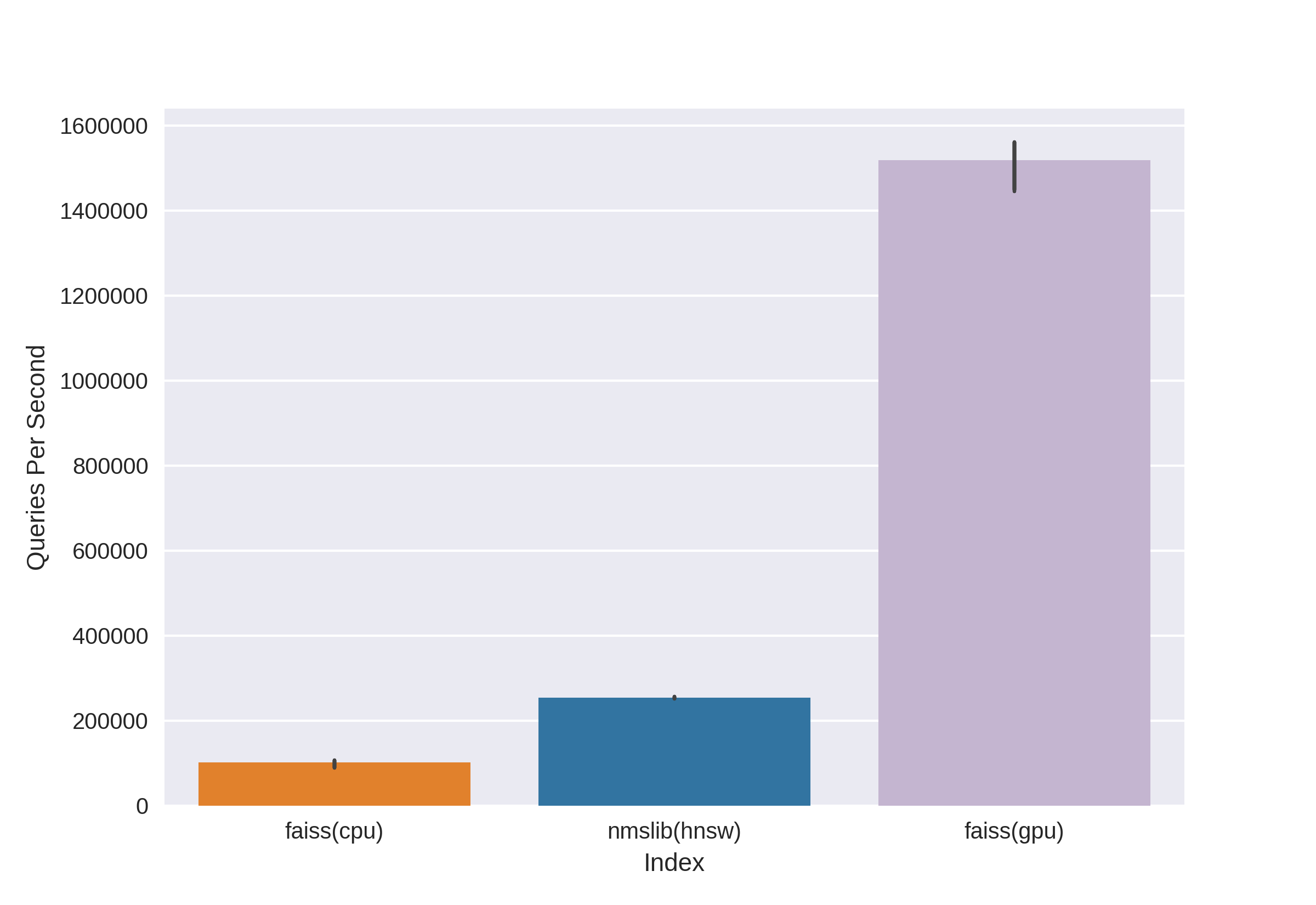
Existen tecnologías conocidas y probadas, como Annoy, FAISS, HNSW. Algoritmo de búsqueda rápida de vecinos HNSW disponible en las bibliotecas nmslib y hnswlib, muestra resultados de vanguardia en la CPU, que se pueden ver desde los mismos puntos de referencia. Pero lo cortamos de inmediato, ya que no estamos contentos con la cantidad de memoria utilizada cuando trabajamos con grandes cantidades de datos. Comenzamos a elegir entre Annoy y FAISS y al final elegimos FAISS por conveniencia, menor uso de memoria, uso potencial en la GPU y puntos de referencia para el rendimiento (puede ver, por ejemplo, aquí ). Por cierto, en FAISS, el algoritmo HNSW se implementa como una opción.
¿Qué es FAISS?
Facebook AI Research Similarity Search es un desarrollo del equipo de Facebook AI Research para encontrar rápidamente vecinos cercanos y agruparlos en el espacio vectorial. La alta velocidad de búsqueda permite trabajar con datos muy grandes, hasta varios miles de millones de vectores.
La principal ventaja de FAISS es sus resultados de vanguardia en la GPU, mientras que su implementación en la CPU es ligeramente inferior a hnsw (nmslib). Queríamos poder buscar tanto en la CPU como en la GPU. Además, FAISS está optimizado en términos de uso de memoria y búsqueda en grandes lotes.
Source
FAISS le permite encontrar rápidamente los k vectores más cercanos para un vector dado x. Pero, ¿cómo funciona esta búsqueda bajo el capó?
Índices
El concepto principal en FAISS es el índice y, de hecho, es solo una colección de parámetros y vectores. Los conjuntos de parámetros son completamente diferentes y dependen de las necesidades del usuario. Los vectores pueden permanecer sin cambios, pero pueden reconstruirse. Algunos índices están disponibles para trabajar inmediatamente después de agregarles vectores, y algunos requieren capacitación previa. Los nombres de los vectores se almacenan en el índice: ya sea en la numeración de 0 a n, o como un número que se ajusta al tipo Int64.
El primer índice, y el más simple que utilizamos en la conferencia, es Flat . Solo almacena todos los vectores en sí mismo, y la búsqueda de un vector dado se realiza mediante una búsqueda exhaustiva, por lo que no hay necesidad de entrenarlo (pero más información sobre el entrenamiento a continuación). En una pequeña cantidad de datos, un índice tan simple puede cubrir sus necesidades de búsqueda.
Ejemplo:
import numpy as np
dim = 512 # 512
nb = 10000 #
nq = 5 #
np.random.seed(228)
vectors = np.random.random((nb, dim)).astype('float32')
query = np.random.random((nq, dim)).astype('float32')
Cree un índice plano y agregue vectores sin capacitación:
import faiss
index = faiss.IndexFlatL2(dim)
print(index.ntotal) #
index.add(vectors)
print(index.ntotal) # 10 000
Ahora busquemos los 7 vecinos más cercanos para los primeros cinco vectores de vectores:
topn = 7
D, I = index.search(vectors[:5], topn) # : Distances, Indices
print(I)
print(D)
Salida
[[0 5662 6778 7738 6931 7809 7184]
[1 5831 8039 2150 5426 4569 6325]
[2 7348 2476 2048 5091 6322 3617]
[3 791 3173 6323 8374 7273 5842]
[4 6236 7548 746 6144 3906 5455]]
[[ 0. 71.53578 72.18823 72.74326 73.2243 73.333244 73.73317 ]
[ 0. 67.604805 68.494774 68.84221 71.839905 72.084335 72.10817 ]
[ 0. 66.717865 67.72709 69.63666 70.35903 70.933304 71.03237 ]
[ 0. 68.26415 68.320595 68.82381 68.86328 69.12087 69.55179 ]
[ 0. 72.03398 72.32417 73.00308 73.13054 73.76181 73.81281 ]]Vemos que los vecinos más cercanos con una distancia de 0 son los propios vectores, el resto se alinean al aumentar la distancia. Busquemos nuestros vectores desde la consulta:
D, I = index.search(query, topn)
print(I)
print(D)
Salida
[[2467 2479 7260 6199 8640 2676 1767]
[2623 8313 1500 7840 5031 52 6455]
[1756 2405 1251 4136 812 6536 307]
[3409 2930 539 8354 9573 6901 5692]
[8032 4271 7761 6305 8929 4137 6480]]
[[73.14189 73.654526 73.89804 74.05615 74.11058 74.13567 74.443436]
[71.830215 72.33813 72.973885 73.08897 73.27939 73.56996 73.72397 ]
[67.49588 69.95635 70.88528 71.08078 71.715965 71.76285 72.1091 ]
[69.11357 69.30089 70.83269 71.05977 71.3577 71.62457 71.72549 ]
[69.46417 69.66577 70.47629 70.54611 70.57645 70.95326 71.032005]]Ahora las distancias en la primera columna de los resultados no son cero, ya que los vectores de consulta no están en el índice.
El índice puede guardarse en el disco y luego cargarse desde el disco:
faiss.write_index(index, "flat.index")
index = faiss.read_index("flat.index")¡Parece que todo es elemental! Algunas líneas de código, y ya tenemos una estructura para buscar vectores de alta dimensión. Pero dicho índice con solo diez millones de vectores de dimensión 512 pesará alrededor de 20 GB y ocupará la misma cantidad de RAM cuando lo use.
En el proyecto para la conferencia, utilizamos un enfoque tan básico con un índice plano, todo fue genial debido a la cantidad relativamente pequeña de datos, ¡pero ahora estamos hablando de decenas y cientos de millones de vectores de alta dimensión!
Acelere su búsqueda con listas invertidas

Fuente
La característica principal y más genial de FAISS es el índice de FIV o elíndice de archivo invertido . La idea de los archivos invertidos es lacónica, y está bellamente explicada con los dedos :
imaginemos un ejército gigantesco que consta de los guerreros más multicolores, que suman, digamos, 1,000,000 de personas. Dirigir todo un ejército a la vez será imposible. Como es habitual en los asuntos militares, necesitamos dividir nuestro ejército en subunidades. Dividámonos enpartes aproximadamente iguales por comandantes en el papel de representante de cada división. E intentaremos enviar los más similares en carácter, origen, datos físicos, etc. guerreros en una unidad, y elegiremos al comandante para que represente a su unidad con la mayor precisión posible: es alguien "promedio". Como resultado, nuestra tarea se redujo de comandar un millón de soldados a comandar 1000 unidades a través de sus comandantes, y tenemos una excelente idea de la composición de nuestro ejército, ya que sabemos qué son los comandantes. Esta es la idea detrás del índice de FIV: agrupemos un gran conjunto de vectores pieza por pieza usando el algoritmok-means
, establecer cada parte de acuerdo con el centroide, es un vector que es el centro seleccionado para un grupo dado. La búsqueda se llevará a cabo a través de la distancia mínima al centroide, y solo entonces buscaremos las distancias mínimas entre los vectores en el grupo que corresponde al centroide dado. Tomando k igual , donde es el número de vectores en el índice, obtendremos una búsqueda óptima en dos niveles, el primero entre centroide, luego entre vectores en cada grupo. La búsqueda es mucho más rápida que la búsqueda de fuerza bruta, que resuelve uno de nuestros problemas cuando trabajamos con muchos millones de vectores. El espacio vectorial se divide por el método k-medias en k grupos. A cada grupo se le asigna un centroide Código de ejemplo:

dim = 512
k = 1000 # “”
quantiser = faiss.IndexFlatL2(dim)
index = faiss.IndexIVFFlat(quantiser, dim, k)
vectors = np.random.random((1000000, dim)).astype('float32') # 1 000 000 “”
Y puede escribirlo de manera mucho más elegante, utilizando la práctica herramienta FAISS para crear el índice:
index = faiss.index_factory(dim, “IVF1000,Flat”)
:
print(index.is_trained) # False.
index.train(vectors) # Train
# , , :
print(index.is_trained) # True
print(index.ntotal) # 0
index.add(vectors)
print(index.ntotal) # 1000000
Después de considerar este tipo de índice después de Flat, resolvimos uno de nuestros problemas potenciales: la velocidad de búsqueda, que se vuelve varias veces más lenta en comparación con una búsqueda completa.
D, I = index.search(query, topn)
print(I)
print(D)
Salida
[[19898 533106 641838 681301 602835 439794 331951]
[654803 472683 538572 126357 288292 835974 308846]
[588393 979151 708282 829598 50812 721369 944102]
[796762 121483 432837 679921 691038 169755 701540]
[980500 435793 906182 893115 439104 298988 676091]]
[[69.88127 71.64444 72.4655 72.54283 72.66737 72.71834 72.83057]
[72.17552 72.28832 72.315926 72.43405 72.53974 72.664055 72.69495]
[67.262115 69.46998 70.08826 70.41119 70.57278 70.62283 71.42067]
[71.293045 71.6647 71.686615 71.915405 72.219505 72.28943 72.29849]
[73.27072 73.96091 74.034706 74.062515 74.24464 74.51218 74.609695]]
Pero hay un "pero": la precisión de la búsqueda, así como la velocidad, dependerán de la cantidad de clústeres visitados, que se pueden configurar con el parámetro nprobe:
print(index.nprobe) # 1 –
index.nprobe = 16 # -16 top-n
D, I = index.search(query, topn)
print(I)
print(D)
Salida
[[ 28707 811973 12310 391153 574413 19898 552495]
[540075 339549 884060 117178 878374 605968 201291]
[588393 235712 123724 104489 277182 656948 662450]
[983754 604268 54894 625338 199198 70698 73403]
[862753 523459 766586 379550 324411 654206 871241]]
[[67.365585 67.38003 68.17187 68.4904 68.63618 69.88127 70.3822]
[65.63759 67.67015 68.18429 68.45782 68.68973 68.82755 69.05]
[67.262115 68.735535 68.83473 68.88733 68.95465 69.11365 69.33717]
[67.32007 68.544685 68.60204 68.60275 68.68633 68.933334 69.17106]
[70.573326 70.730286 70.78615 70.85502 71.467674 71.59512 71.909836]]Como puede ver, después de aumentar nprobe, tenemos resultados completamente diferentes, la parte superior de las distancias más pequeñas en D mejoró.
Puede tomar nprobe igual al número de centroides en el índice, entonces esto será equivalente a buscar por búsqueda exhaustiva, la precisión será mayor, pero la velocidad de búsqueda disminuirá notablemente.
Búsqueda en el disco: listas invertidas en disco
¡Genial, resolvimos el primer problema, ahora tenemos una velocidad de búsqueda aceptable en decenas de millones de vectores! Pero todo esto es inútil siempre que nuestro enorme índice no se ajuste a la RAM.
Específicamente para nuestra tarea, la principal ventaja de FAISS es la capacidad de almacenar Listas Invertidas del índice de FIV en el disco, cargando solo metadatos en la RAM.
¿Cómo creamos dicho índice? Entrenamos indexIVF con los parámetros necesarios sobre la cantidad máxima posible de datos que cabe en la memoria, luego agregamos vectores (aquellos que han sido entrenados y no solo) al índice entrenado en partes y escribimos el índice para cada una de las partes en el disco.
index = faiss.index_factory(512, “,IVF65536, Flat”, faiss.METRIC_L2)Entrenamos el índice en la GPU de esta manera:
res = faiss.StandardGpuResources()
index_ivf = faiss.extract_index_ivf(index)
index_flat = faiss.IndexFlatL2(512)
clustering_index = faiss.index_cpu_to_gpu(res, 0, index_flat) # 0 – GPU
index_ivf.clustering_index = clustering_indexfaiss.index_cpu_to_gpu (res, 0, index_flat) se puede reemplazar con faiss.index_cpu_to_all_gpus (index_flat) para usar todas las GPU juntas.
Es altamente deseable que la muestra de entrenamiento sea lo más representativa posible y tenga una distribución uniforme, por lo que precomponemos el conjunto de datos de entrenamiento del número requerido de vectores, seleccionándolos aleatoriamente de todo el conjunto de datos.
train_vectors = ... #
index.train(train_vectors)
# , :
faiss.write_index(index, "trained_block.index")
#
# :
for bno in range(first_block, last_block+ 1):
block_vectors = vectors_parts[bno]
block_vectors_ids = vectors_parts_ids[bno] # id ,
index = faiss.read_index("trained_block.index")
index.add_with_ids(block_vectors, block_vectors_ids)
faiss.write_index(index, "block_{}.index".format(bno))
Después de eso, combinamos todas las Listas Invertidas juntas. Esto es posible, ya que cada uno de los bloques es esencialmente el mismo índice entrenado, solo que con diferentes vectores dentro.
ivfs = []
for bno in range(first_block, last_block+ 1):
index = faiss.read_index("block_{}.index".format(bno), faiss.IO_FLAG_MMAP)
ivfs.append(index.invlists)
# index inv_lists
# :
index.own_invlists = False
# :
index = faiss.read_index("trained_block.index")
# invlists
# invlists merged_index.ivfdata
invlists = faiss.OnDiskInvertedLists(index.nlist, index.code_size, "merged_index.ivfdata")
ivf_vector = faiss.InvertedListsPtrVector()
for ivf in ivfs:
ivf_vector.push_back(ivf)
ntotal = invlists.merge_from(ivf_vector.data(), ivf_vector.size())
index.ntotal = ntotal #
index.replace_invlists(invlists)
faiss.write_index(index, data_path + "populated.index") #
En pocas palabras: ahora nuestro índice es populated.index y archivos merged_blocks.ivfdata .
En populated.index registró la ruta completa original al archivo con las Listas Invertidas, por lo que si la ruta del archivo ivfdata por alguna razón, el cambio en la lectura del índice necesitará usar el indicador faiss.IO_FLAG_ONDISK_SAME_DIR , que le permite buscar el archivo ivfdata en el mismo directorio que el populated.index:
index = faiss.read_index('populated.index', faiss.IO_FLAG_ONDISK_SAME_DIR)
El ejemplo de demostración del proyecto Github FAISS se tomó como base .
Puede encontrar una mini guía para elegir un índice en el Wiki de FAISS . Por ejemplo, pudimos colocar un conjunto de datos de entrenamiento de 12 millones de vectores en RAM, por lo que elegimos el índice IVFFlat en 262144 centroides y luego lo escalamos a cientos de millones. La guía también propone usar el índice IVF262144_HNSW32, en el que el vector que pertenece al clúster está determinado por el algoritmo HNSW con 32 vecinos más cercanos (en otras palabras, se usa el cuantificador IndexHNSWFlat), pero, como nos pareció durante otras pruebas, la búsqueda de dicho índice es menos precisa. Además, debe tenerse en cuenta que dicho cuantificador excluye la posibilidad de usar en la GPU.
Revelación:
Uso de disco significativamente reducido con la cuantificación del producto
Gracias al método de búsqueda de disco, fue posible eliminar la carga de la RAM, pero un índice con un millón de vectores todavía tomó alrededor de 2 GB de espacio en disco, y estamos hablando de la posibilidad de trabajar con miles de millones de vectores, ¡lo que requeriría más de dos TB! Por supuesto, el volumen no es tan grande si establece un objetivo y asigna espacio de disco adicional, pero nos molestó un poco.
Y aquí la codificación vectorial viene al rescate, a saber, la cuantificación escalar (SQ) y la cuantificación del producto (PQ). SQ: codifica cada componente del vector en n bits (generalmente 8, 6 o 4 bits). Consideraremos la opción PQ, porque la idea de codificar un solo componente de tipo float32 con ocho bits parece demasiado deprimente en términos de precisión. Aunque en algunos casos la compresión de SQfp16 a float16 tendrá una precisión casi sin pérdidas.
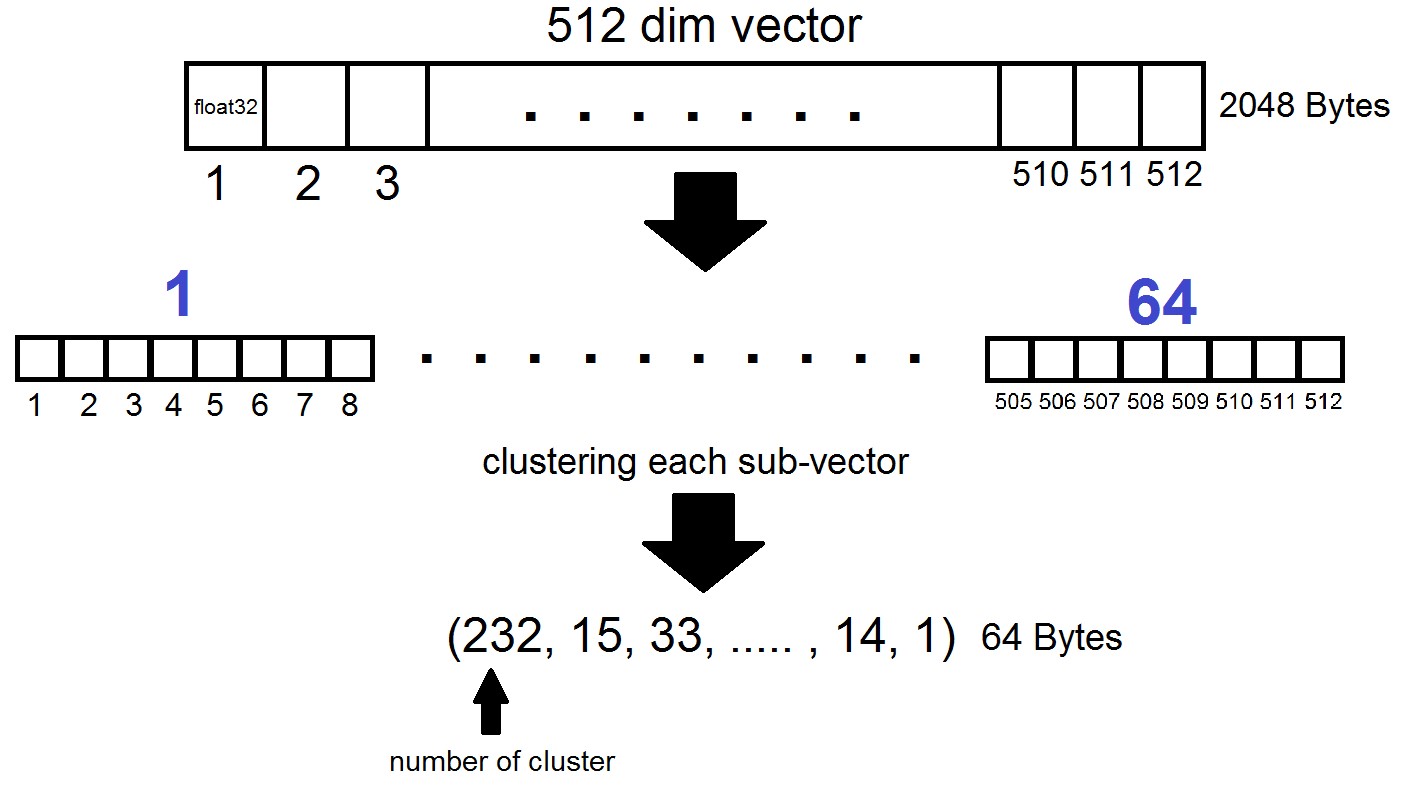
La esencia de la cuantificación del producto es la siguiente: los vectores de dimensión 512 se dividen en n partes, cada una de las cuales se agrupa en 256 grupos posibles (1 byte), es decir, representamos un vector con n bytes, donde n es generalmente como máximo 64 en una implementación FAISS. ¡Pero tal cuantización se aplica no a los vectores del conjunto de datos en sí, sino a las diferencias de estos vectores y los centroides correspondientes obtenidos durante la generación de Listas Invertidas! Resulta que las Listas Invertidas serán conjuntos codificados de distancias entre vectores y sus centroides.
index = faiss.index_factory(dim, "IVF262144,PQ64", faiss.METRIC_L2)Resulta que ahora no es necesario que almacenemos todos los vectores: es suficiente asignar n bytes por vector y 2048 bytes por cada vector centroide. En nuestro caso, tomamos , es decir: la longitud de un subvector, que se define en uno de los 256 grupos. Al buscar por el vector x, los centroides más cercanos se determinarán primero con el cuantificador plano habitual, y luego x también se divide en sub-vectores, cada uno de los cuales está codificado por el número de uno de los 256 centroides correspondientes. Y la distancia al vector se define como la suma de 64 distancias entre los sub-vectores.
Cual es el resultado?
Detuvimos nuestros experimentos en el índice "IVF262144, PQ64", ya que satisfizo completamente todas nuestras necesidades de velocidad y precisión de búsqueda, y también aseguró el uso razonable del espacio en disco con una mayor escala del índice. Más específicamente, en este momento, con 315 millones de vectores, el índice ocupa 22 GB de espacio en disco y aproximadamente 3 GB de RAM cuando se usa.
Otro detalle interesante que no mencionamos anteriormente es la métrica utilizada por el índice. Por defecto, las distancias entre dos vectores se consideran en la métrica euclidiana L2, o en un lenguaje más comprensible, las distancias se consideran como la raíz cuadrada de la suma de los cuadrados de las diferencias de coordenadas. Pero puede establecer otra métrica, en particular, probamos la métrica METRIC_INNER_PRODUCT, o la métrica de las distancias del coseno entre vectores. Es coseno porque el coseno del ángulo entre dos vectores en el sistema de coordenadas euclidiana se expresa como la relación del producto escalar (en coordenadas) al producto de sus longitudes, y si todos los vectores en nuestro espacio tienen longitud 1, entonces el coseno del ángulo será exactamente igual al producto en forma de coordenadas. En este caso, cuanto más cerca estén los vectores en el espacio, más cerca de uno estará su producto de punto.
La métrica L2 tiene una transición matemática directa a la métrica del producto punto. Sin embargo, al comparar experimentalmente las dos métricas, la impresión fue que la métrica de los productos escalares nos ayuda a analizar los coeficientes de similitud de las imágenes de una manera más exitosa. Además, las incrustaciones de nuestras fotos se obtuvieron utilizandoInsightFace , que implementa la arquitectura ArcFace utilizando distancias de coseno. También hay otras métricas en los índices FAISS que puede leer aquí .
algunas palabras sobre GPU
Conclusión y ejemplos interesantes.
Entonces, de vuelta a donde todo comenzó. Y comenzó, recuerde, con la motivación de resolver el problema de encontrar bots en la red de Instagram y, más específicamente, buscar publicaciones duplicadas con personas o avatares en ciertos grupos de usuarios. En el proceso de redacción del material, quedó claro que una descripción detallada de nuestra metodología de búsqueda de bots se dibuja en un artículo separado, que discutiremos en futuras publicaciones, pero por ahora nos limitaremos a ejemplos de nuestros experimentos con FAISS.
Puede vectorizar imágenes o caras de diferentes maneras, elegimos la tecnología InsightFace (vectorizar imágenes y seleccionar características n-dimensionales de ellas es una larga historia separada). En el curso de los experimentos con la infraestructura que recibimos, se descubrieron propiedades bastante interesantes y divertidas.
Por ejemplo, con el permiso de colegas y conocidos, subimos sus rostros en una búsqueda y rápidamente encontramos las fotos en las que están presentes:

nuestro colega se metió en la foto de un visitante de la Comic-Con, estando en segundo plano en la multitud. Fuente

Picnic en un gran grupo de amigos, foto de la cuenta de un amigo. Fuente

Simplemente pasando. Un fotógrafo desconocido capturó a los chicos por su perfil temático. No sabían dónde había terminado su foto, y después de 5 años olvidaron por completo cómo fueron fotografiados. Fuente

En este caso, el fotógrafo es desconocido y fotografiado en secreto.
Inmediatamente recordé a una chica sospechosa con una cámara réflex, sentada enfrente en ese momento :) Fuente
Por lo tanto, mediante acciones simples, FAISS le permite ensamblar un análogo del conocido FindFace en su rodilla.
Otra característica interesante: en el índice FAISS, cuanto más se parecen las caras entre sí, más cerca están los vectores correspondientes en el espacio. Decidí mirar más de cerca los resultados de búsqueda un poco menos precisos en mi cara y encontré clones terriblemente similares a mí :)

Algunos de los clones del autor.
Fuentes fotográficas: 1 , 2 , 3
En general, FAISS abre un campo enorme para la implementación de cualquier idea creativa. Por ejemplo, usando el mismo principio de proximidad vectorial de caras similares, uno podría construir caminos de una persona a otra. O, como último recurso, haga de FAISS una fábrica para la producción de tales memes:

Fuente ¡
Gracias por su atención y esperamos que este material sea útil para los lectores de Habr!
Este artículo fue escrito con el apoyo de mis colegas Artyom Korolyov (korolevart), Timur Kadyrov y Arina Reshetnikova.
I + D Dentsu Aegis Network Rusia.