El experimento de secuenciación del transcriptoma (RNA-seq) se ha convertido casi en un procedimiento de rutina para estudiar tanto organismos modelo como cultivos. Como resultado del procesamiento bioinformático de dichos experimentos, se obtienen datos volumétricos heterogéneos, representados por las secuencias de nucleótidos de las transcripciones, secuencias de aminoácidos y su anotación estructural y funcional. Es importante presentar los datos obtenidos a una amplia gama de investigadores en forma de bases de datos (DB). En esta publicación, consideraremos un enfoque híbrido para la creación de bases de datos de genética molecular que contienen información sobre secuencias de transcripciones y sus anotaciones estructurales y funcionales. La esencia del enfoque es el almacenamiento simultáneo de datos estructurados y débilmente estructurados en la base de datos.La tecnología se utiliza para implementar una base de datos de transcriptomas de plantas agrícolas. La publicación analiza las características de la implementación de este enfoque y ejemplos de la formación de consultas simples y complejas a dicha base de datos en SQL. Este artículo es un resumen abreviado de nuestro trabajo doi: 10.17537 / 2020.15.455 en el que soy coautor.
Introducción
El estudio de transcriptomas vegetales mediante secuenciación de alto rendimiento (secuenciación de ARN, RNA-seq) se utiliza ampliamente en la actualidad para resolver problemas como evaluar la expresión génica para diferentes genotipos y en diferentes condiciones ambientales, identificar secuencias de ARN (para organismos no modelo), buscando marcadores para genes funcionalmente importantes. El experimento RNA-seq se ha convertido casi en un procedimiento de rutina para estudiar ambos organismos modelo ( Arabidopsis thaliana) y para cultivos agrícolas (tomate, maíz, cebada, trigo, etc.). Los resultados de un experimento de transcriptoma son pequeños fragmentos de secuencias de nucleótidos, y solo el procesamiento bioinformático, que incluye varias etapas, permite obtener secuencias de transcripción y su anotación funcional en base a ellas. Son los resultados del procesamiento bioinformático los que interesan a un biólogo y pueden ser interpretados en términos de las funciones de los genes, sus productos, niveles de expresión, variaciones genéticas, etc. Cabe señalar que como resultado de un experimento de transcriptoma, se obtiene una gran cantidad de datos (decenas y cientos de miles de secuencias), acceso gratuito y conveniente al cual es importante proporcionar a una amplia gama de biólogos.lejos del procesamiento bioinformático rutinario de los resultados de la secuenciación. Este propósito es servido por bases de datos que tienen una interfaz de usuario conveniente y organizan enlaces entre secuencias biológicas y su anotación funcional. Dichas bases de datos incluyen el Atlas de expresión del Instituto Europeo de Bioinformática, EGENES, una base de datos de información sobre vías metabólicas de genes basada en datos de transcriptomas, una base de datos sobre expresión génica para ciertos tipos de organismos: TodoFirgene para abeto Abies sachalinensis; atlas de expresión génica para rosa; base de datos de transcriptomas anotados de pino costero EuroPineDB, etc.Dichas bases de datos incluyen el Atlas de expresión del Instituto Europeo de Bioinformática, EGENES, una base de datos de información sobre vías metabólicas de genes basada en datos de transcriptomas, una base de datos sobre expresión génica para ciertos tipos de organismos: TodoFirgene para abeto Abies sachalinensis; atlas de expresión génica para rosa; la base de datos de transcriptomas anotados de pino costero EuroPineDB, etc.Dichas bases de datos incluyen el Atlas de expresión del Instituto Europeo de Bioinformática, EGENES, una base de datos de información sobre vías metabólicas de genes basada en datos de transcriptomas, una base de datos sobre expresión génica para ciertos tipos de organismos: TodoFirgene para abeto Abies sachalinensis; atlas de expresión génica para rosa; la base de datos de transcriptomas anotados de pino costero EuroPineDB, etc.
RNA-seq , , : , , (, , , .), , , . . ( FASTA, FASTQ), ( BAM, SAM, PSL ..) (BED, GFF, GTF). ( TSV, XLS). RDBMS ( , . Relational DataBase Management System): , , ..
, , , RNA-seq , . , . , . , . , - : , , . , . NoSQL ( SQL, . not only SQL).
, RDBMS NoSQL , (, , , ). , , , , , . : , , . , , . OORT (Out Of Reference Transcripts), , , , : , , , . 1241 20440228 4055996 . OORT .
OORT :
;
, de novo ;
, , ;
( , , );
( , ).
, , , , , . , . , .
,
OORT PostgreSQL 12. , , (. “ ”). .
Python c (. “ OORT”) “”, JSON SQLAlchemy. SQLAlchemy Python SQL , Python. JSONB .
, ( ), . , . , , int, . (int, real, text) B-tree, , , “”, “”, “”. B-tree .
, , . (GO) ; ; .. JSON: - . PostgreSQL 12 JSON JSONB. JSON, , SQL. JSONB , , , . JSON GIN . , , .
“ ”, , PostgreQSL, [41]. , “ ”, . ( ) , , .
OORT SQL. , , :
SELECT
– . , , * .
FROM
– . , . JOIN, .
WHERE
– . AND, OR NOT ( ).
, PostgreSQL 12 SQL:2016 , JSONB. '->'
, JSON- , '->>'
, . “ ”. SQL OORT , DataGrip, pgAdmin psql PostgreSQL.
de novo : (study), (exp), (contig) (pep) (. 1). “ , ”, , :
id, ;
exp study_id, study;
contig exp_id, exp;
pep contig_id exp_id, contig exp, .
, , , . OORT 1.
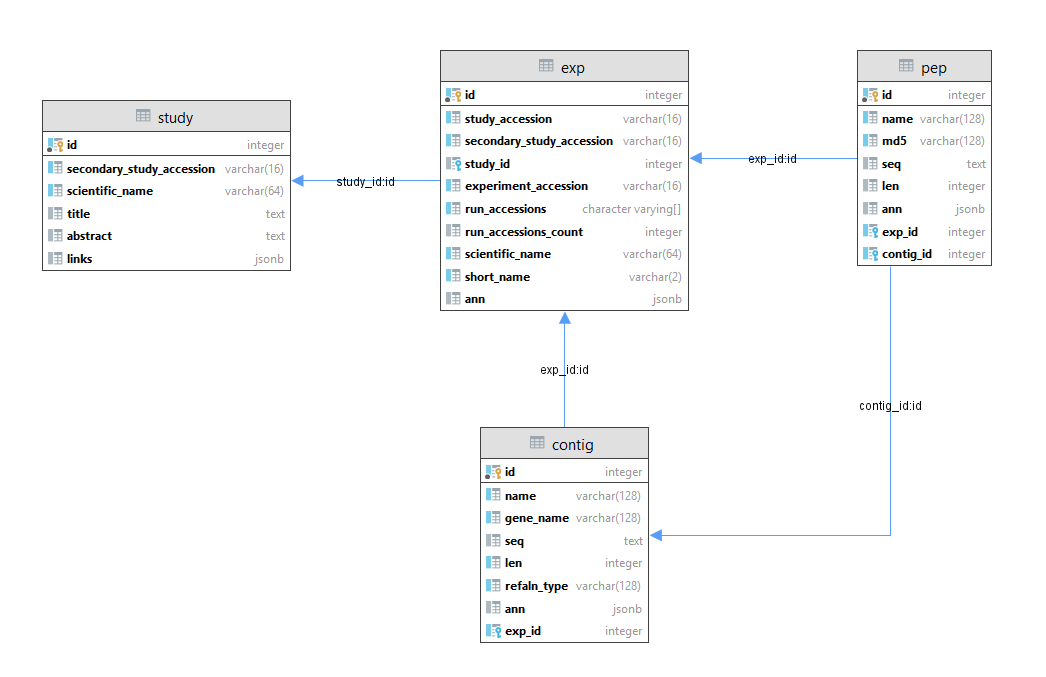
OORT . 45.5 .
|
|
|
|
2766 |
3.0 |
|
1241 |
7.4 |
|
20440228 |
39000.0 |
|
4055996 |
6495.0 |
OORT. ‘exp’, ‘pep’ ‘contig’ jsonb, . TRINITY_DN3631_c0_g1_i1 kallisto JSON ‘ann’ ‘contig’ OORT:
"kallisto": {
"g": {
"tpm": 8.44,
"eff_length": 341.61,
"est_counts": 35.0
},
"i": {
"tpm": 8.43596,
"eff_length": 341.614,
"est_counts": 35.0
}
}
‘ann’ ‘contig’ TRINITY_DN3631_c0_g1_i1, , kallisto. ‘ann’ “kallisto” ( ). kallisto , ( , “g”, ), , ( , “i”, ). kallisto : “tpm”, " ”, TPM; “eff_length”, ; “est_counts”, , . , JSON.
. Interproscan :
tsv – InterproScan;
go_ann – GO ;
pathways_ann – , .
TransDecoder :
type – (, -, );
chain – , (“+” “–”);
score – , ;
cds_end – : ;
cds_start – : .
PostgreSQL 12 OORT. , (O. sativa). SQL :
-- 1: 'Oryza sativa'
select contig.seq
from contig join exp on exp.id = contig.exp_id
where scientific_name = 'Oryza sativa'
, 3 7 TPM. JSON ‘ann’ “kallisto” ->
->>
.
-- 2: , 3.0 7.0 TPM
select *
from contig
where ((ann -> 'kallisto' -> 'g' ->> 'tpm')::real)
between 3.0 and 7.0
, (.. - md5), .
-- 3: ,
--
select subseq.exp_id, subseq.study_id,
max(subseq.contig_g_tpm) as contig_g_tpm,
study.scientific_name, tissue_type
from (
select (contig.ann->'kallisto'->'g'->'tpm')::real
as contig_g_tpm, contig.id as contig_id, pep.id as
pep_id, contig.exp_id, e.study_id, pep.md5,
e.ann->'ebi'->'tissue_type' as tissue_type
from pep join contig on pep.contig_id = contig.id
join exp e on contig.exp_id = e.id
where pep.md5 = '857c1634328c5333ab73efcc11a45038'
)
subseq join study on subseq.study_id = study.id
group by exp_id, study_id, study.scientific_name,
tissue_type
4 , , InterproScan. , GIN ‘ann’ ‘pep’ “go_ann”. .
-- 4:
--
-- c
create index ix_pep_ann_interpro_go_ann on pep using
gin (((ann -> 'interpro'::text) -> 'go_ann'::text))
--
select * from pep
where (ann -> 'interpro' -> 'go_ann')
?& array['GO:0055085', 'GO:0006811']
5 , OORT, ‘Categorizing’. GIN ‘abstract’ (4 , to_tsvector), . ( 9–10).
-- 5:
--
create index study_abstract_idx on study
(to_tsvector('english'::regconfig, abstract));
--
select *
from study
where to_tsvector(study.abstract)
@@ plainto_tsquery('Categorizing')
6 , , gmap. “gmap” , “ ” , ( 2–9). ( 12–15) ( 19–30).
-- contig_gmap_view_sqlalchemy
create materialized view
contig_gmap_view_sqlalchemy as
select db.id, jsonb_array_elements(
db.ann -> 'gmap'::text) AS gmap
from (
select contig.id, contig.ann
from contig ORDER BY contig.id
) db;
-- GMAP
create index gmap_start_end_chr_idx
on contig_gmap_view_sqlalchemy
(((gmap ->> 15)::integer), ((gmap ->> 16)::integer),
(gmap ->> 13));
-- , 2 2000 5000
--
select pep.id AS pep_id
from exp join contig ON exp.id = contig.exp_id
join contig_gmap_view_sqlalchemy
on contig.id = contig_gmap_view_sqlalchemy.id
join pep ON pep.contig_id = contig.id
where
cast(contig_gmap_view_sqlalchemy.gmap ->> 15
as integer) >=2000 AND
cast(contig_gmap_view_sqlalchemy.gmap ->> 16
as integer) <= 5000
and(contig_gmap_view_sqlalchemy.gmap ->> 13) = '2'
and exp.scientific_name like 'Zea mays'
, , , , . , .
- , () , , . , : , , , , JSON.
. , JSON- . , , . JSON : JSON , \ -, .
- , - . , . : , , JSON, .
. , , doi: 10.17537/2020.15.455.