Buen día, queridos Habradams y Habragospoda. En este artículo cerraremos las escotillas de nuestro batiscafo lo más herméticamente posible, añadiremos velocidad a nuestro motor Python y nos sumergiremos en el abismo de las estadísticas, a una profundidad en la que la luz del sol prácticamente no penetra. A esta profundidad, encontraremos una gran cantidad de todo tipo de pruebas estadísticas que flotan ante nosotros en forma de fórmulas extrañas. Al principio nos parecerá que todos están ordenados de forma diferente, pero intentaremos llegar al fondo del principal motor impulsor de todas estas extrañas criaturas.
¿De qué debo advertirle antes de bucear a esta profundidad? Primero, supongo que ya ha leído el libro "Estadísticas para todos" de Sarah Boslaf, y también ha buscado en la documentación oficial del módulo de estadísticas de la biblioteca SciPy. Perdóname por mi próxima suposición, pero me parece que lo más probable es que te quedaras un poco estupefacto por la gran cantidad de pruebas que existen, y te quedaste estupefacto aún más cuando te diste cuenta de que esto es solo la punta del iceberg. Bueno, si aún no ha encontrado todos los placeres de esta maravillosa "pubertad", le recomiendo que obtenga el libro de Alexander Ivanovich Kobzar "Estadística matemática aplicada. Para ingenieros y científicos". Bueno, si estás "en el tema", entonces mira debajo del gato,¿Por qué? Porque la presentación e interpretación de los hechos es a veces más importante e interesante que los hechos mismos.
, :
import numpy as np
import pandas as pd
from scipy import stats
import matplotlib.pyplot as plt
import seaborn as sns
from pylab import rcParams
sns.set()
rcParams['figure.figsize'] = 10, 6
%config InlineBackend.figure_format = 'svg'
np.random.seed(42)
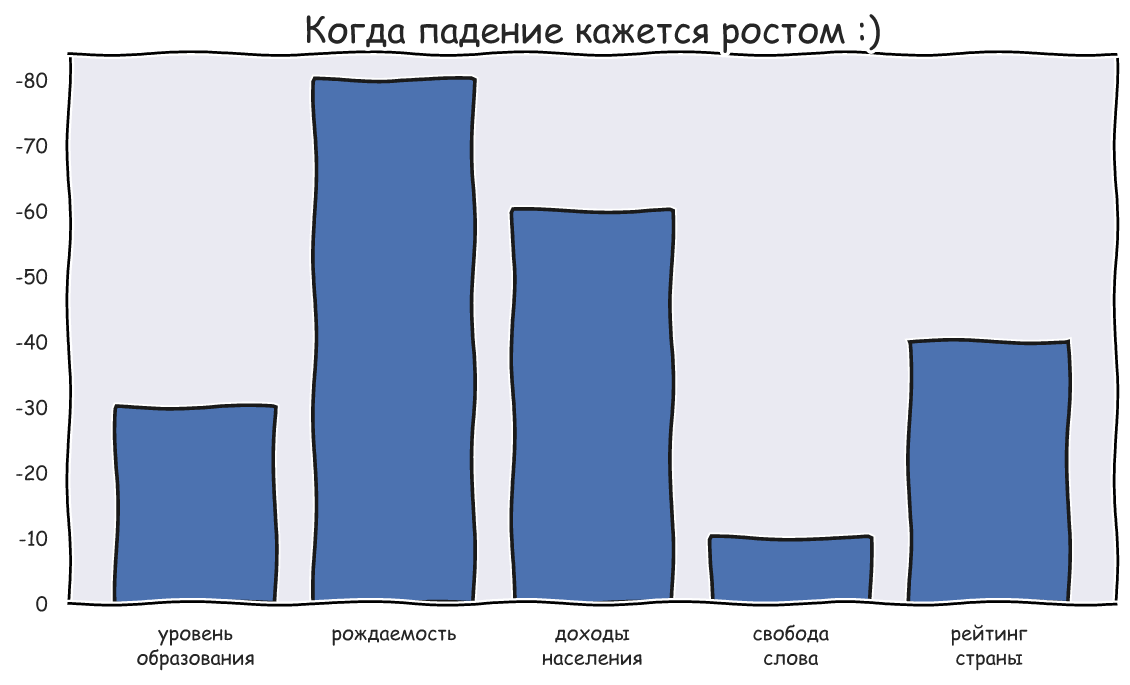
, , . , , - , , . [0; 10] 0 - "", 10 - " ". , :
- , - , - . :
, , , . , . , . , .. . " Pthon - " -.
, , . " , , ", - , , . , : , , .. .. , :
" " ;
" " ;
" " .
, . , , . :
x1 = np.array([7.68,5.40,3.99,3.27,2.70,5.85,6.53,5.00,4.60,6.18])
x2 = np.array([1.33,1.66,2.76,4.56,4.75,0.70,3.13,1.96,4.60,3.69])
fig, ax = plt.subplots()
sns.ecdfplot(x=x1, ax=ax, label=' ')
sns.ecdfplot(x=x2, ax=ax, label='')
ax.legend();
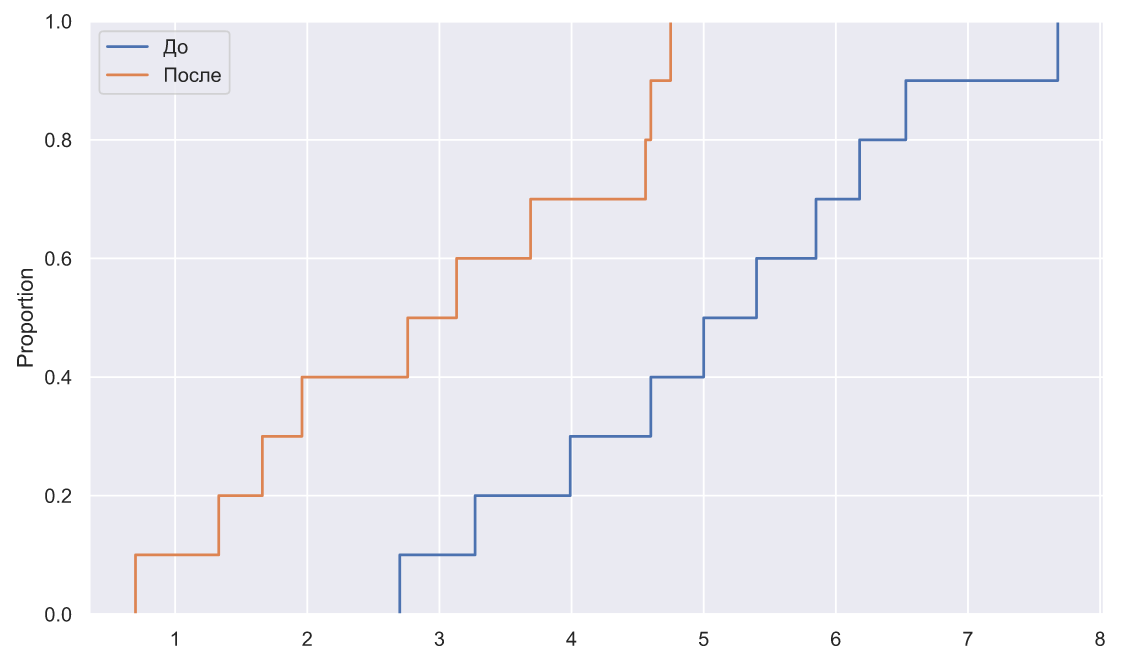
, , ( ), , :
plt.bar(np.arange(10), (x2-x1));
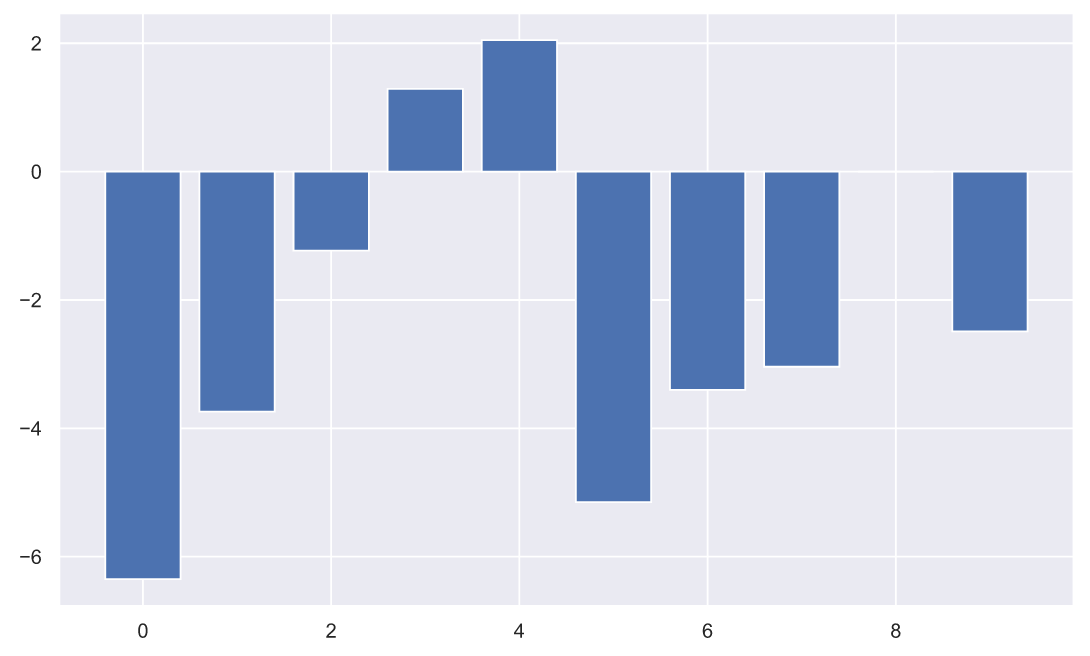
, - . , , , . , :
plt.bar(np.arange(10), (x2-x1))
plt.xticks(np.arange(10));
plt.title(' ( - )',
fontsize=15)
plt.xlabel('id ', fontsize=15)
plt.ylabel('');
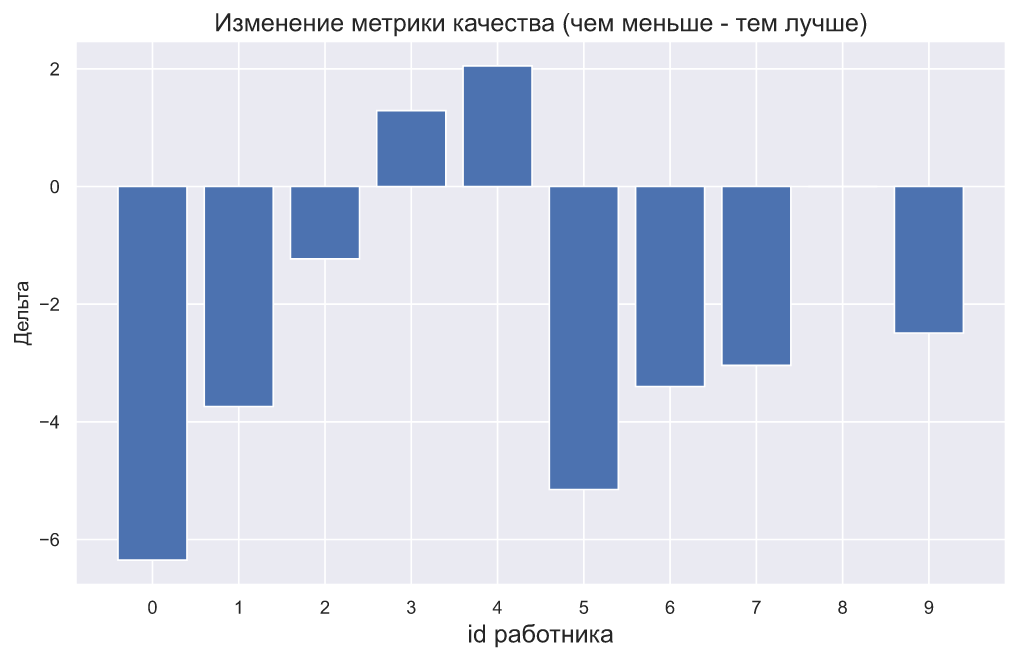
:
plt.bar(np.arange(10) - 0.2, x1, width=0.4, label='')
plt.bar(np.arange(10) + 0.2, x2, width=0.4, label='')
plt.xticks(np.arange(10))
plt.legend()
plt.title(' ( - )',
fontsize=15)
plt.xlabel('id ', fontsize=15)
plt.ylabel('');
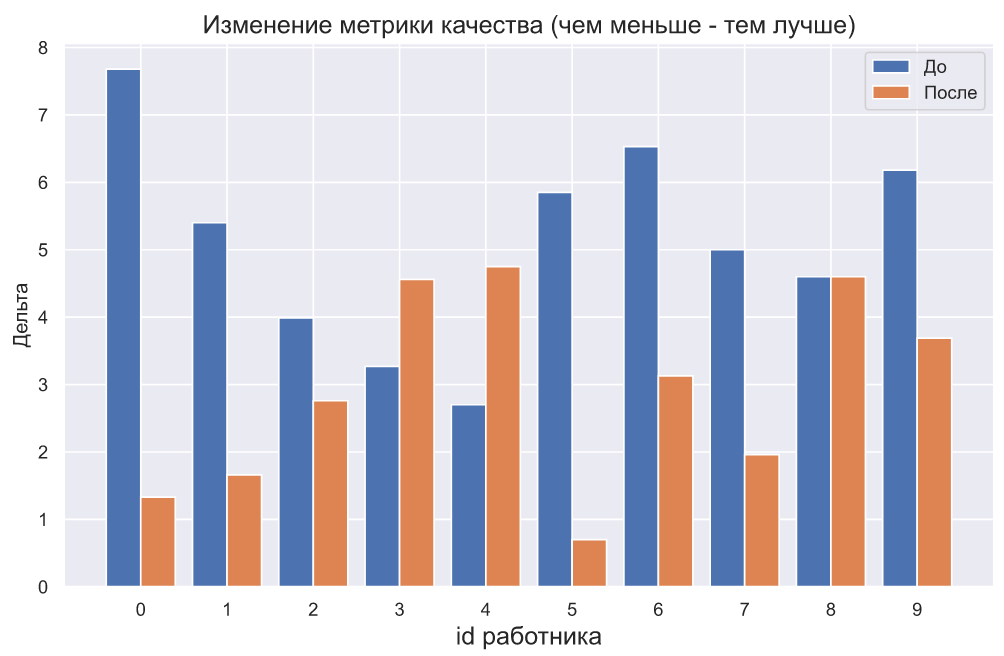
, - , , , . , . , , , , . - .
, - t- :
stats.ttest_rel(x2, x1)
Ttest_relResult(statistic=-2.5653968678354184, pvalue=0.03041662395965993)
c p-value 0.03 , . , . - . ?
:
print(f'mean(x1) = {x1.mean():.3}')
print(f'mean(x2) = {x2.mean():.3}')
print('-'*15)
print(f'std(x1) = {x1.std(ddof=1):.3}')
print(f'std(x2) = {x2.std(ddof=1):.3}')
mean(x1) = 5.12
mean(x2) = 2.91
---------------
std(x1) = 1.53
std(x2) = 1.47
t- , , (, ), . ? , ? ? , , .. , . , ?
( ) - -. , () , . , - . - , . , , - - . - , .
, t- , , . , . , . , ?
, . 5000
10 , :
samples = stats.norm.rvs(loc=(5, 3), scale=1.5, size=(5000, 10, 2))
deviations = samples.var(axis=1, ddof=1)
deviations_df = pd.DataFrame(deviations, columns=['s1', 's2'])
sns.histplot(data=deviations_df, element="poly", color='r', fill=False);
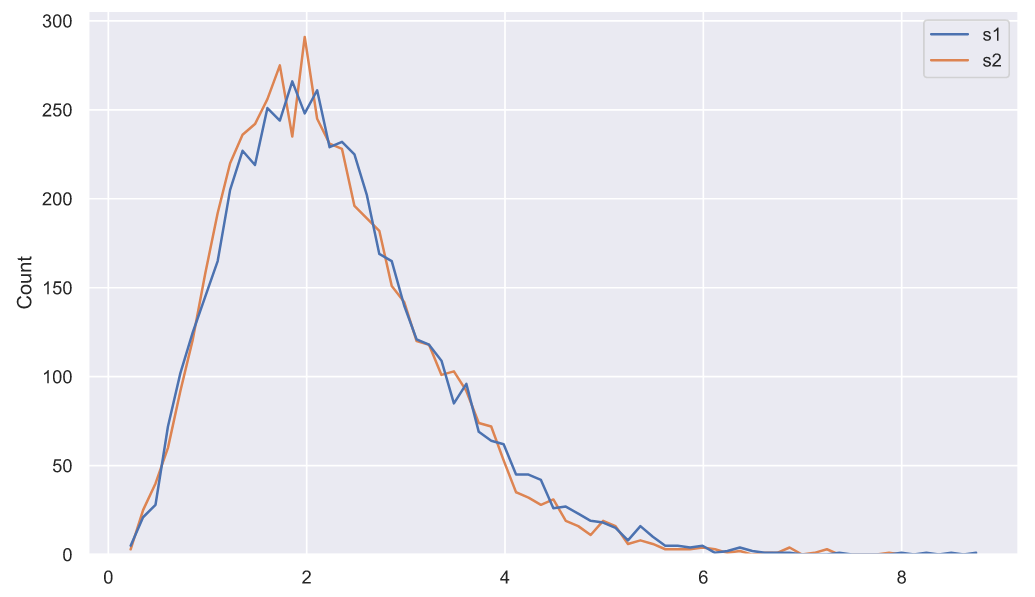
, , "" - . :
sns.histplot(data=pd.DataFrame(np.std(stats.norm.rvs(loc=(5, 3), scale=1.5, size=(5000,10,2)), axis=1, ddof=1), columns=['s1', 's2']), element="poly", color='r', fill=False);
"" - . - , , :
- ;
;
, .
, - "", "". " ", .. . , , , . , .
, . , , , . : "" . , ", " - , - . , fit():
df1, loc1, scale1 = stats.chi2.fit(deviations_df['s1'], fdf=10)
print(f'df1 = {df1}, loc1 = {loc1:<8.4}, scale1 = {scale1:.3}')
df2, loc2, scale2 = stats.chi2.fit(deviations_df['s2'], fdf=10)
print(f'df2 = {df2}, loc2 = {loc2:<8.4}, scale1 = {scale2:.3}')
df1 = 10, loc1 = -0.1027 , scale1 = 0.238
df2 = 10, loc2 = -0.08352, scale1 = 0.231
fig, ax = plt.subplots()
# ,
# 0.5 1:
sns.histplot(data=deviations_df['s1'], color='r', element='poly',
fill=False, stat='density', label='s1', ax=ax)
sns.histplot(data=deviations_df['s2'], color='b', element='poly',
fill=False, stat='density', label='s2', ax=ax)
chi2_rv1 = stats.chi2(df1, loc1, scale1)
chi2_rv2 = stats.chi2(df2, loc2, scale2)
x = np.linspace(0, 8, 300)
sns.lineplot(x=x, y=chi2_rv1.pdf(x), color='r', label='pdf(s1)', ax=ax)
sns.lineplot(x=x, y=chi2_rv2.pdf(x), color='b', label='pdf(s2)', ax=ax)
ax.set_xticks(np.arange(10))
ax.set_xlabel('s');
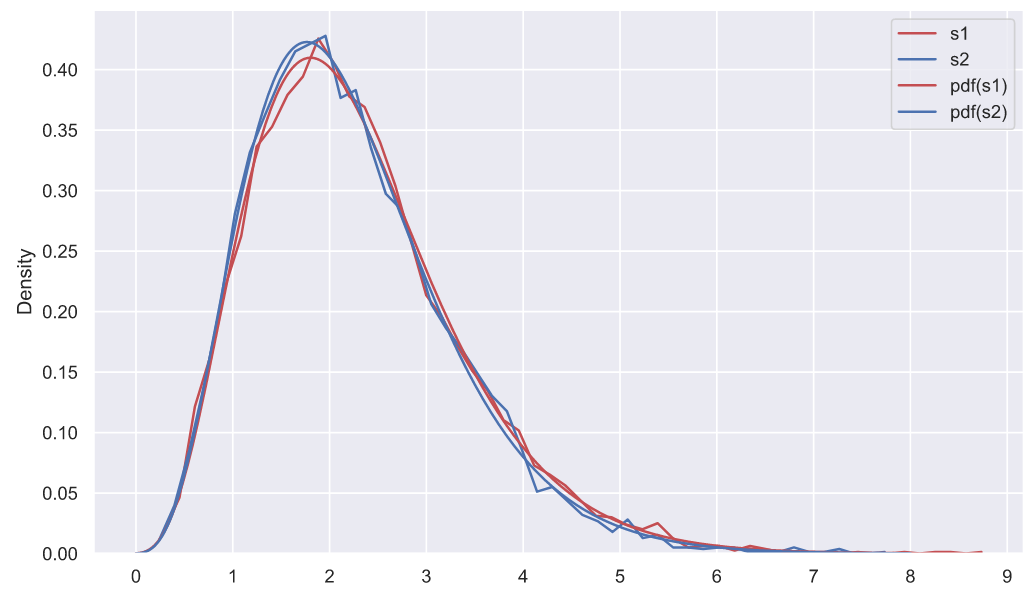
- . , , , , , , ( ). . , (""), ("") ("") (""), , , . , , , "" , , .
? , ,
. , . ? , 2,
?
fig, ax = plt.subplots()
var = 2**2
x = np.linspace(0, 10, 300)
sns.lineplot(x=x, y=chi2_rv1.pdf(x), label='pdf(s1)', ax=ax)
ax.vlines(var, 0, chi2_rv1.pdf(var), color='r', lw=2)
ax.fill_between(x[x>var], chi2_rv1.pdf(x[x>var]),
np.zeros(len(x[x>var])), alpha=0.3, color='b')
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
p = chi2_rv1.sf(var)
ax.set_title(f'$P(s_{1} \geqslant {var}) = $' + '{:.3}'.format(p))
ax.set_xlabel('s');
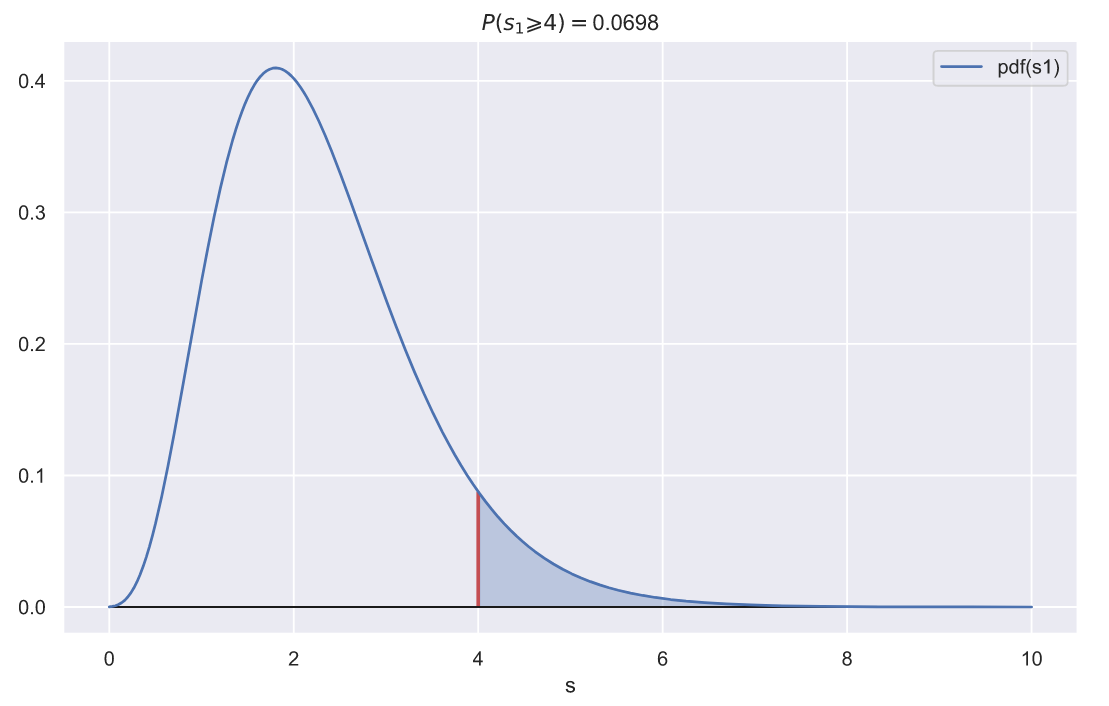
p-value , , , 10
. ,
1.5. ,
, - , .
, - , , 0.1:
fig, ax = plt.subplots()
x = np.linspace(0, 10, 300)
sns.lineplot(x=x, y=chi2_rv1.pdf(x), label='pdf(s1)', ax=ax)
# :
ci_left, ci_right = chi2_rv1.interval(0.9)
ax.vlines([ci_left, ci_right], 0, 0.5, color='r', lw=2)
x_le_ci_l, x_ge_ci_r = x[x<ci_left], x[x>ci_right]
ax.fill_between(x_le_ci_l, chi2_rv1.pdf(x_le_ci_l),
np.zeros(len(x_le_ci_l)), alpha=0.3, color='b')
ax.fill_between(x_ge_ci_r, chi2_rv1.pdf(x_ge_ci_r),
np.zeros(len(x_ge_ci_r)), alpha=0.3, color='b')
ax.set_title(f'P({ci_left:.3} $\geqslant s_{1} \geqslant$ {ci_right:.3}) = 0.1')
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
ax.set_xlabel('s');
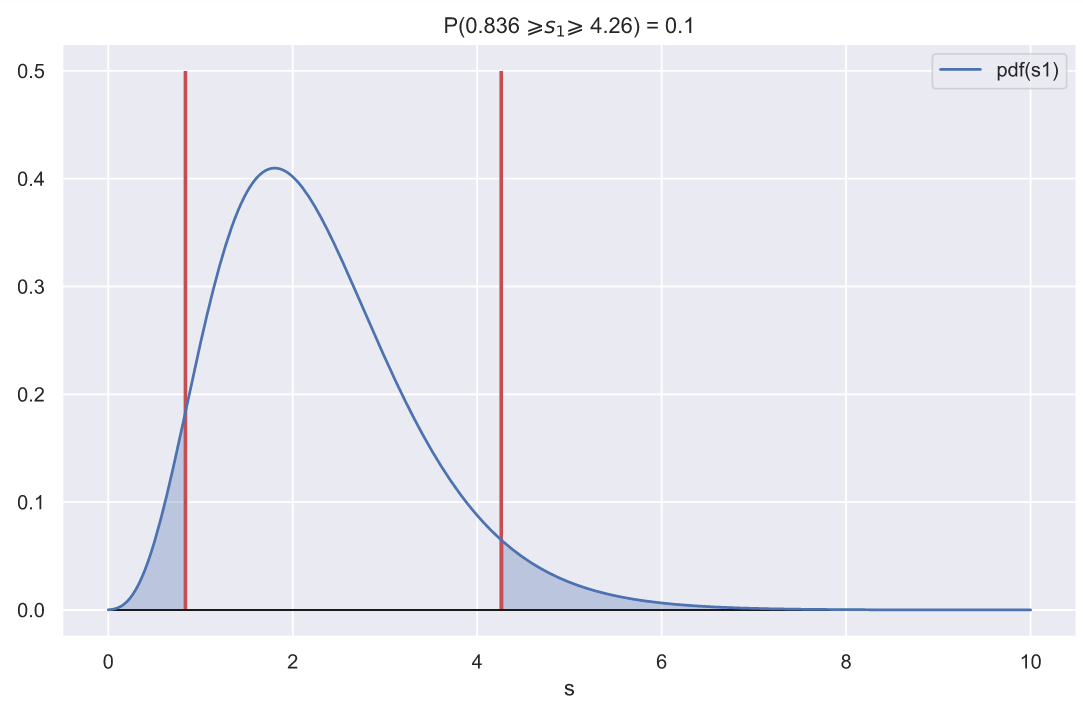
, , - , , .
? , - . , :
:
rel_dev = deviations_df['s1'] / deviations_df['s2']
sns.histplot(x=rel_dev, stat='density');
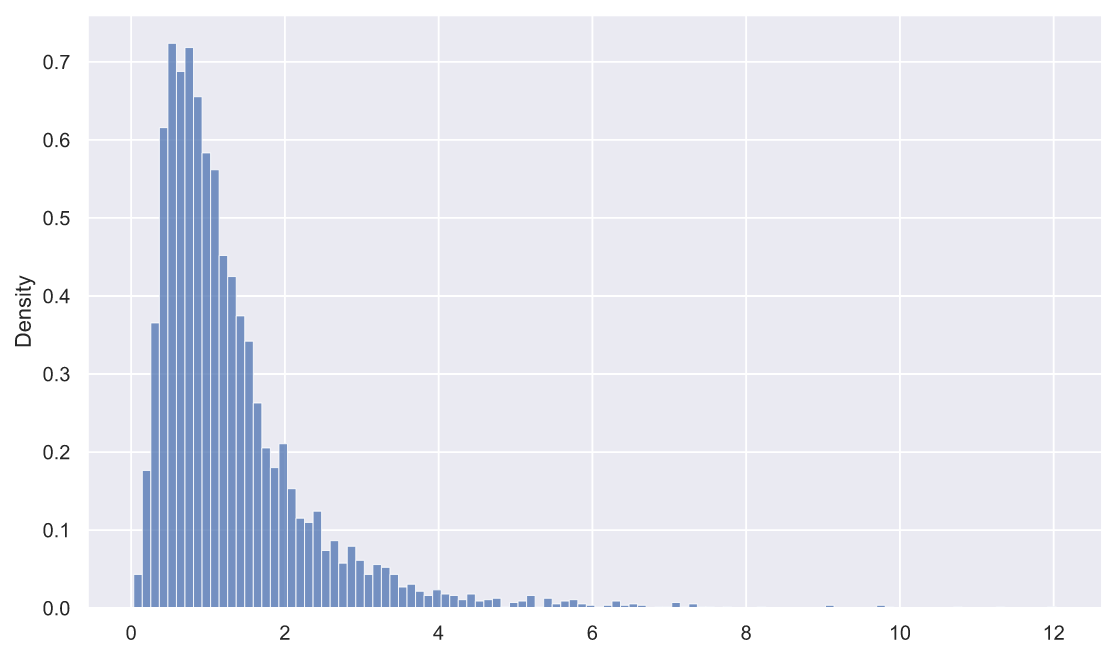
, fit():
dfn, dfd, loc, scale = stats.f.fit(rel_dev, fdfn=10, fdfd=10)
print(f'dfn = {dfn}, dfd = {dfd}, loc2 = {loc2:<8.4}, scale1 = {scale2:.3}')
dfn = 10, dfd = 10, loc2 = -0.08352, scale1 = 0.231
fig, ax = plt.subplots()
rel_dev = deviations_df['s1'] / deviations_df['s2']
sns.histplot(x=rel_dev, stat='density', alpha=0.4)
f_rv = stats.f(dfn, dfd, loc, scale)
x = np.linspace(0, 12, 300)
ax.plot(x, f_rv.pdf(x), color='r')
ax.set_xlim(0, 8);

. , 3, 1, :
fig, ax = plt.subplots()
rel_var = 3
x = np.linspace(0, 8, 300)
sns.lineplot(x=x, y=f_rv.pdf(x), label='pdf(s1)', ax=ax)
ax.vlines(rel_var, 0, f_rv.pdf(rel_var), color='r', lw=2)
ax.fill_between(x[x>rel_var], f_rv.pdf(x[x>rel_var]),
np.zeros(len(x[x>rel_var])), alpha=0.3, color='b')
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
p = f_rv.sf(var)
ax.set_title(f'$P(s_{1}/s_{2} \geqslant {rel_var}) = $' + '{:.3}'.format(p))
ax.set_xlabel('s');
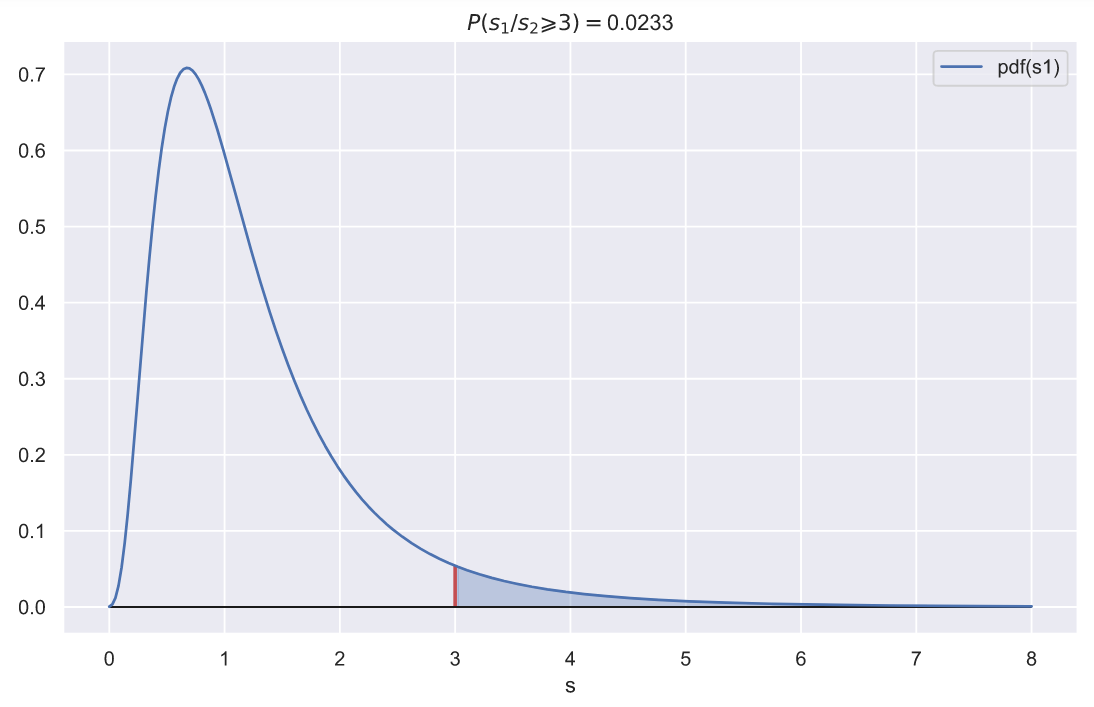
, 10
, , 3, 0.023. , .
:
np.var(x1, ddof=1) / np.var(x2, ddof=1)
1.083553459313125
. , , . , ? ANOVA? , , , , . f_oneway() ( pvalue, , ):
stats.f_oneway(x1, x2)
F_onewayResult(statistic=10.786061383971454, pvalue=0.0041224402038065235)
? - ?
, f_oneway(), :
m1, m2, m = *np.mean((x1, x2), axis=1), np.mean((x1, x2))
ms_bg = (10*(m1 - m)**2 + 10*(m2 - m)**2)/(2 - 1)
ms_wg = (np.sum((x1 - m1)**2) + np.sum((x2 - m2)**2))/(20 - 2)
s = ms_bg/ms_wg
p = stats.f.sf(s, dfn=1, dfd=18)
print(f'statistic = {s:.5}, p-value = {p:.5}')
statistic = 10.786, p-value = 0.0041224
(mean square between group) , . , ,
.
(mean square within group) , , . , , , . , - , , . , ,
:
:
N = 10000
samples_1 = stats.norm.rvs(loc=0, scale=1, size=(N, 10))
samples_2 = stats.norm.rvs(loc=0, scale=1, size=(N, 10))
m1 = samples_1.mean(axis=1)
m2 = samples_2.mean(axis=1)
m = np.hstack((samples_1, samples_2)).mean(axis=1)
ms_bg = 10*((m1 - m)**2 + (m2 - m)**2)
ss_1 = np.sum((samples_1 - m1.reshape(N, 1))**2, axis=1)
ss_2 = np.sum((samples_2 - m2.reshape(N, 1))**2, axis=1)
ms_wg = (ss_1 + ss_2)/18
statistics = ms_bg/ms_wg
f, ax = plt.subplots()
x = np.linspace(0.02, 30, 500)
plt.plot(x, stats.f.pdf(x, dfn=1, dfd=18), color='r', label=f'f.pdf(x, 1, 18, 0, 1)')
plt.legend()
sns.histplot(x=statistics, binwidth=0.1, stat='density', alpha=0.5)
ax.set_xlim(0, 6)
ax.set_title(r' $MS_{bg} \; / \;MS_{wg}$ ');
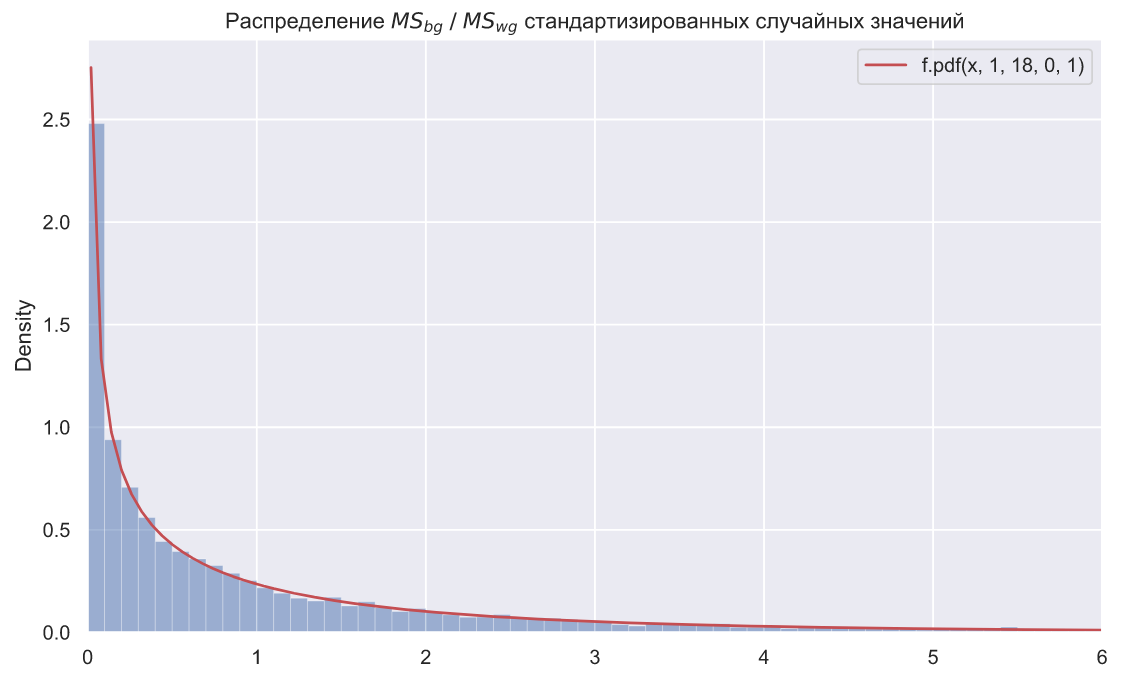
N = 10000
mu_1 = stats.uniform.rvs(loc=0, scale=5, size=(N, 1))
samples_1 = stats.norm.rvs(loc=mu_1, scale=2, size=(N, 10))
mu_2 = stats.uniform.rvs(loc=0, scale=5, size=(N, 1))
samples_2 = stats.norm.rvs(loc=mu_2, scale=2, size=(N, 10))
m1 = samples_1.mean(axis=1)
m2 = samples_2.mean(axis=1)
m = np.hstack((samples_1, samples_2)).mean(axis=1)
ms_bg = 10*((m1 - m)**2 + (m2 - m)**2)
ss_1 = np.sum((samples_1 - m1.reshape(N, 1))**2, axis=1)
ss_2 = np.sum((samples_2 - m2.reshape(N, 1))**2, axis=1)
ms_wg = (ss_1 + ss_2)/18
statistics = ms_bg/ms_wg
loc, scale = stats.f.fit(statistics, fdfn=1, fdfd=18)[-2:]
f, ax = plt.subplots()
x = np.linspace(0.02, 30, 500)
plt.plot(x, stats.f.pdf(x, dfn=1, dfd=18, loc=loc, scale=scale), color='r', label=f'f.pdf(x, 1, 18, {loc:.3}, {scale:.3})')
plt.legend()
sns.histplot(x=statistics, binwidth=0.2, stat='density', alpha=0.5)
ax.set_xlim(0, 20)
ax.set_title(r' $MS_{bg} \; / \;MS_{wg}$ ');
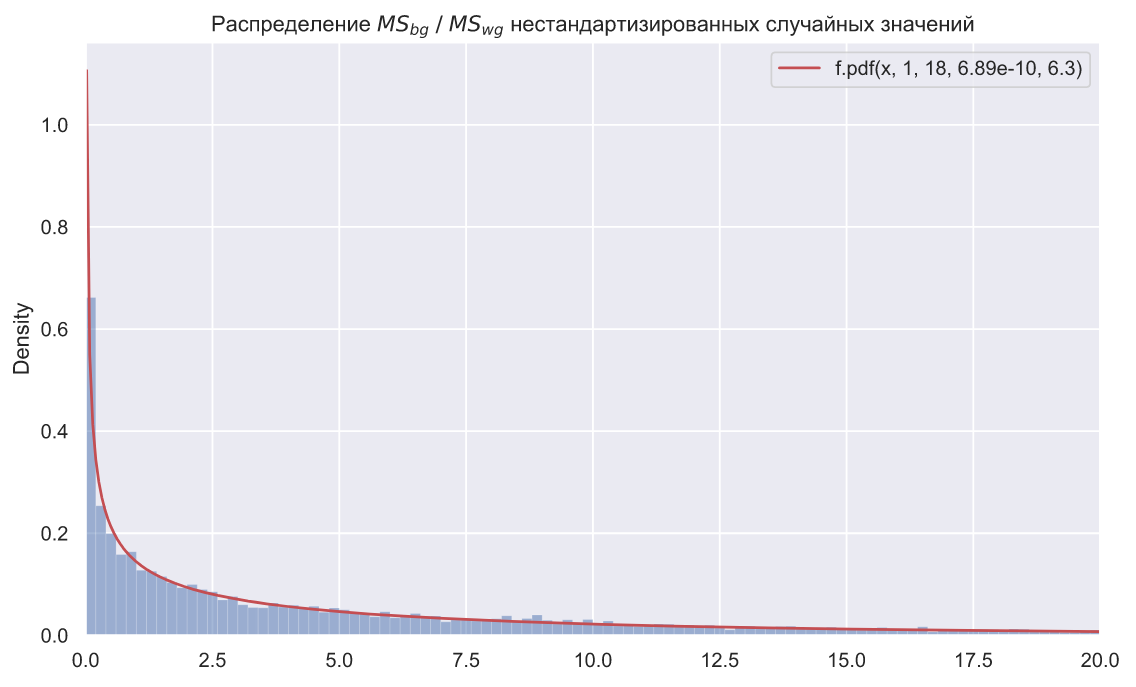
, SciPy levene(). () , ANOVA, :
stats.levene(x1, x2)
LeveneResult(statistic=0.0047521397921121405, pvalue=0.9458007897725039)
""
, , . , , , , , . , , . : 10000 5 , , , :
N, k = 10000, 5
func = [stats.uniform, stats.norm, stats.laplace]
color = list('gyb')
labels = ['s_uniform', 's_norm', 's_laplace']
for i in range(3):
ss = np.square(func[i].rvs(size=(N, k))).sum(axis=1)
sns.histplot(x=ss, stat='density', label=labels[i] + '.pdf(x)',
element='step', color=color[i], lw=2, fill=False)
x = np.linspace(0, 25, 300)
plt.plot(x, stats.chi2.pdf(x, df=5), color='r', label='chi2.pdf(x, df=5)')
plt.legend()
plt.xlim(0, 20);
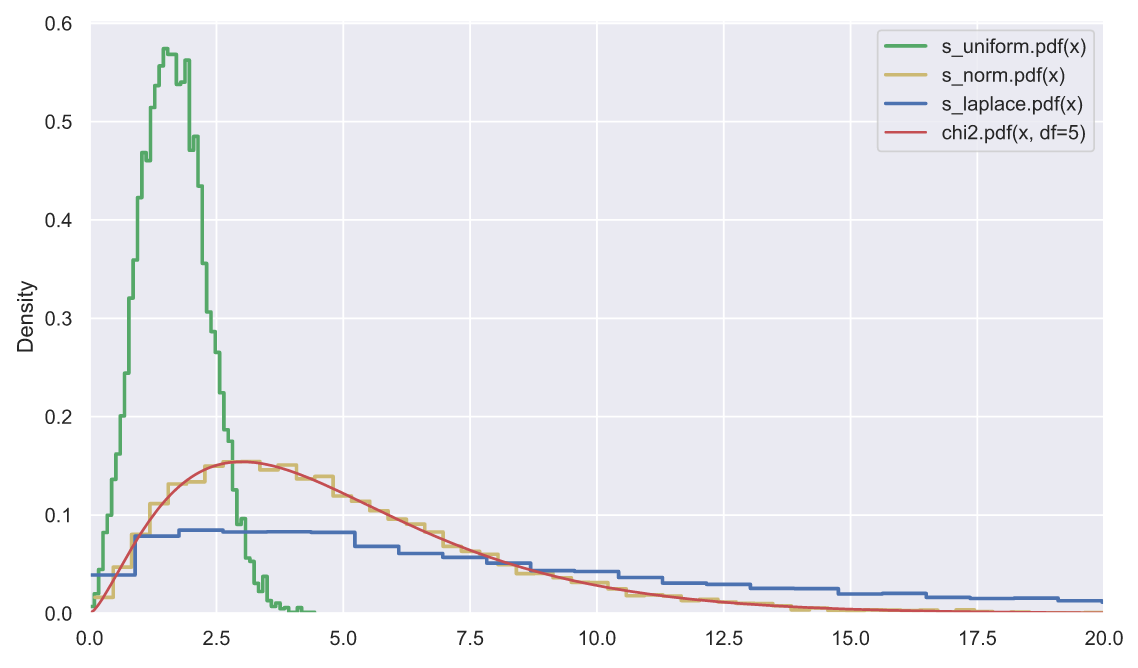
, - , , , "" . , , , - (- - ).
- ANOVA, , "" ? , :
array([0.40572556, 0.67443266, 0.38765587, 0.96540199, 0.57933085])
, - . ? 50 5 , , , :
s = np.sort(stats.norm.rvs(size=(50000, 5)), axis=1).T
for i in range(5):
sns.histplot(x=s[i], stat='density',
label='i = ' + str(i),
element='poly', lw=2, fill=False)
plt.xticks(np.arange(-5, 6))
plt.legend();
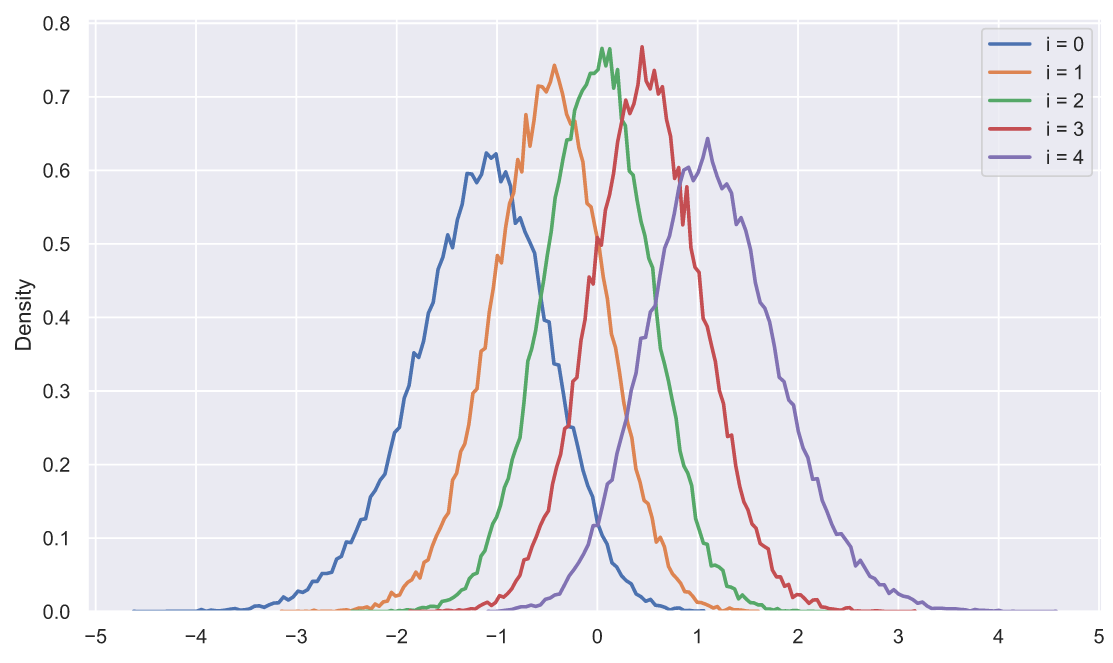
, ? , , , . , :
" " ;
;
( );
- , ( );
.
, , , , "" . ?!! , , - :
s = np.sort(stats.norm.rvs(size=(50000, 5)), axis=1).T
sample = np.sort(stats.norm.rvs(size=5))
colors = sns.color_palette('tab10', 5)
for i in range(5):
sns.histplot(x=s[i], stat='density',
label='i = ' + str(i),
element='poly', lw=2, fill=False,
color=sns.color_palette('pastel', 5)[i])
plt.vlines(sample[i], 0, 0.8, lw=2, zorder=10,
color=sns.color_palette('tab10', 5)[i])
plt.xticks(np.arange(-5, 6))
plt.legend();
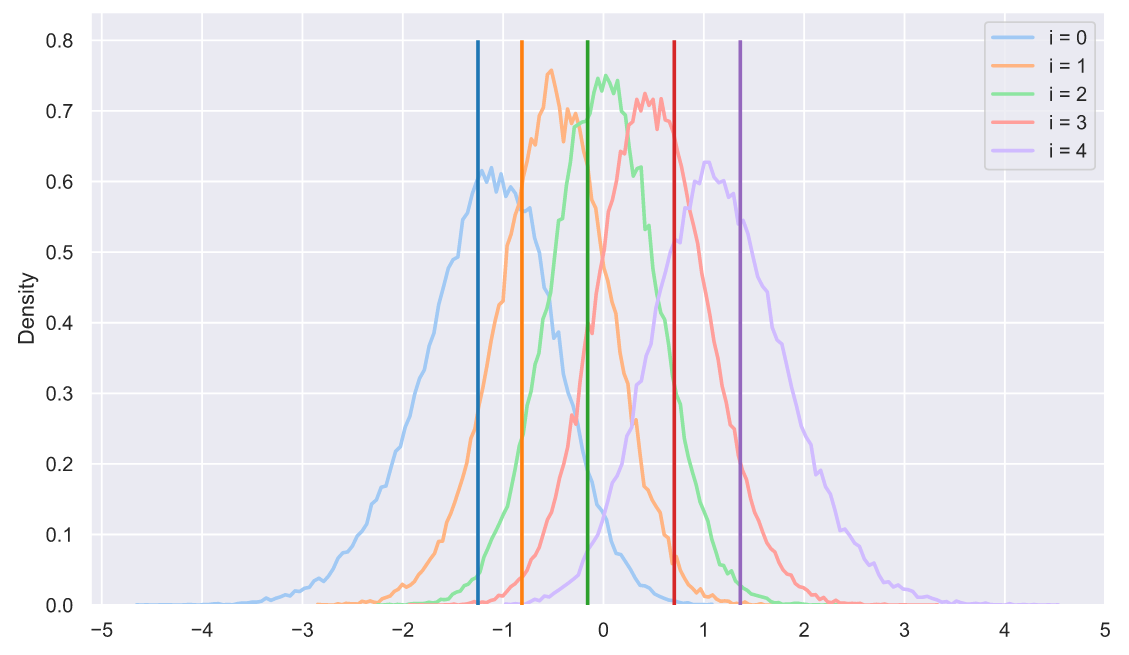
- :
s = np.sort(stats.norm.rvs(size=(50000, 5)), axis=1).T
sample = np.sort(stats.uniform.rvs(loc=-2, scale=4, size=5))
colors = sns.color_palette('tab10', 5)
for i in range(5):
sns.histplot(x=s[i], stat='density',
label='i = ' + str(i),
element='poly', lw=2, fill=False,
color=sns.color_palette('pastel', 5)[i])
plt.vlines(sample[i], 0, 0.8, lw=2, zorder=10,
color=sns.color_palette('tab10', 5)[i])
plt.xticks(np.arange(-5, 6))
plt.legend();
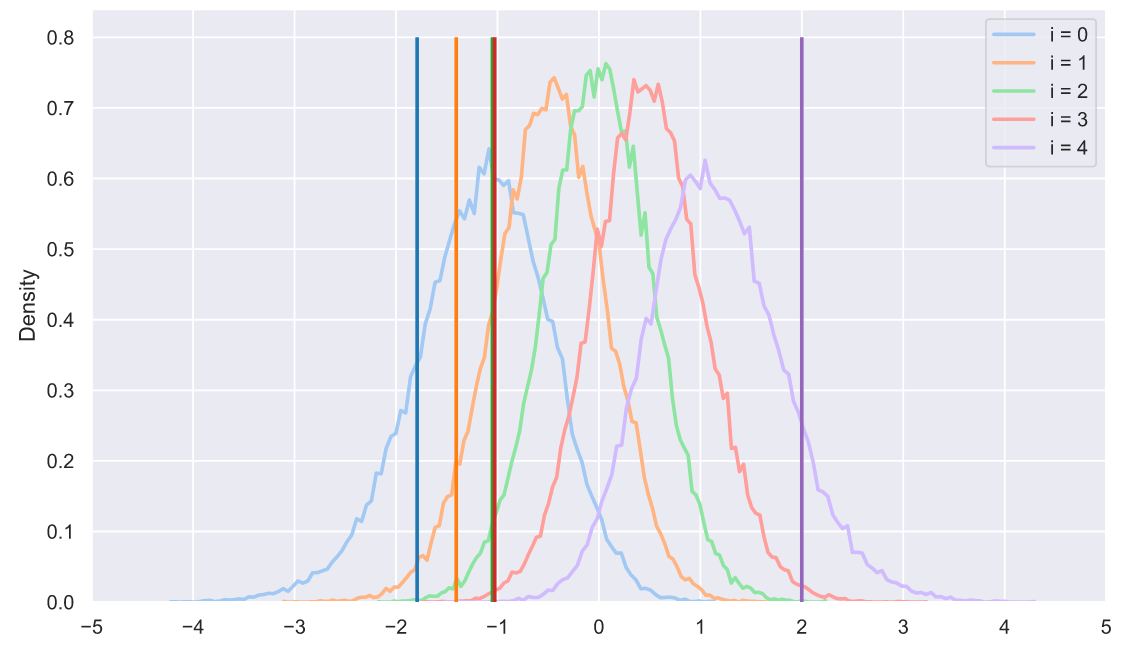
, - . , , - . : , . , . , . :
stats.ks_1samp(x1, stats.norm.cdf, args=(5, 1.5))
KstestResult(statistic=0.11452966409855592, pvalue=0.9971279018404035)
, p-value, , "" . , , . , , :
x1.sort()
n = len(x1)
ecdf_ge = np.r_[1:n+1]/n
ecdf_le = np.r_[0:n]/n
cdf_teor = stats.norm.cdf(x1, loc=5, scale=1.5)
plt.plot(x1, ecdf_ge, color='b', drawstyle='steps-post', label='ecdf_ge')
plt.plot(x1, cdf_teor, color='r', drawstyle='steps-post', label='cdf_teor')
plt.plot(x1, ecdf_le, color='g', drawstyle='steps-post', label='ecdf_le')
plt.legend();
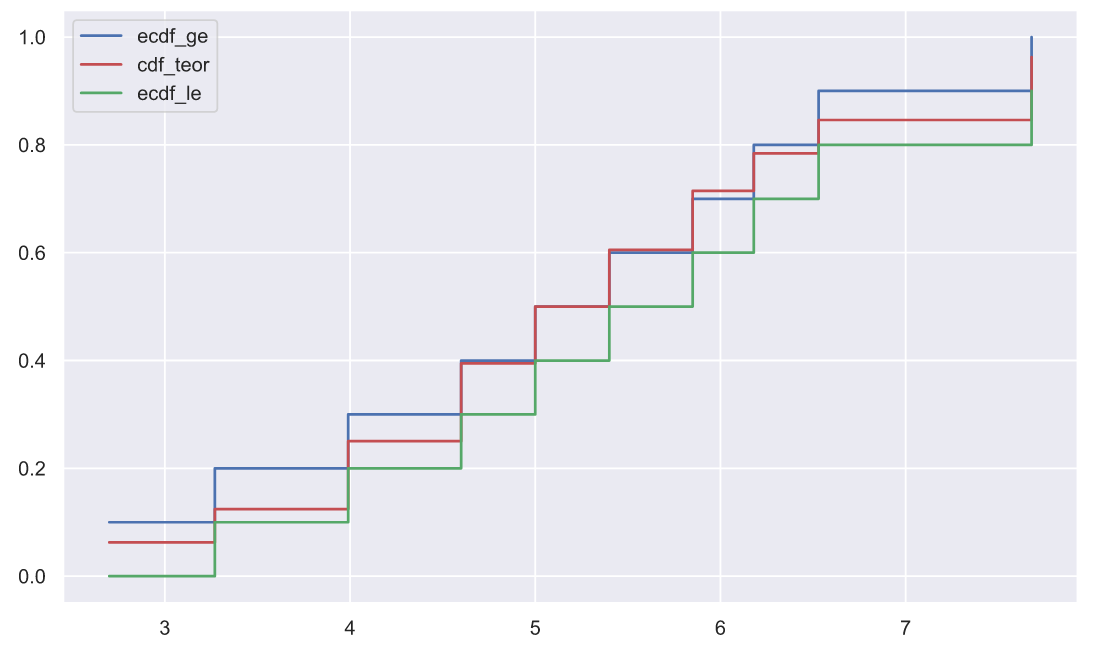
, .. :
d_plus = ecdf_ge - cdf_teor d_minus = cdf_teor - ecdf_le statistic = np.max([d_plus, d_minus]) statistic
0.11452966409855592
( n=5):
x = np.linspace(0, 1, 3000)
plt.plot(x, stats.kstwo.pdf(x, n));
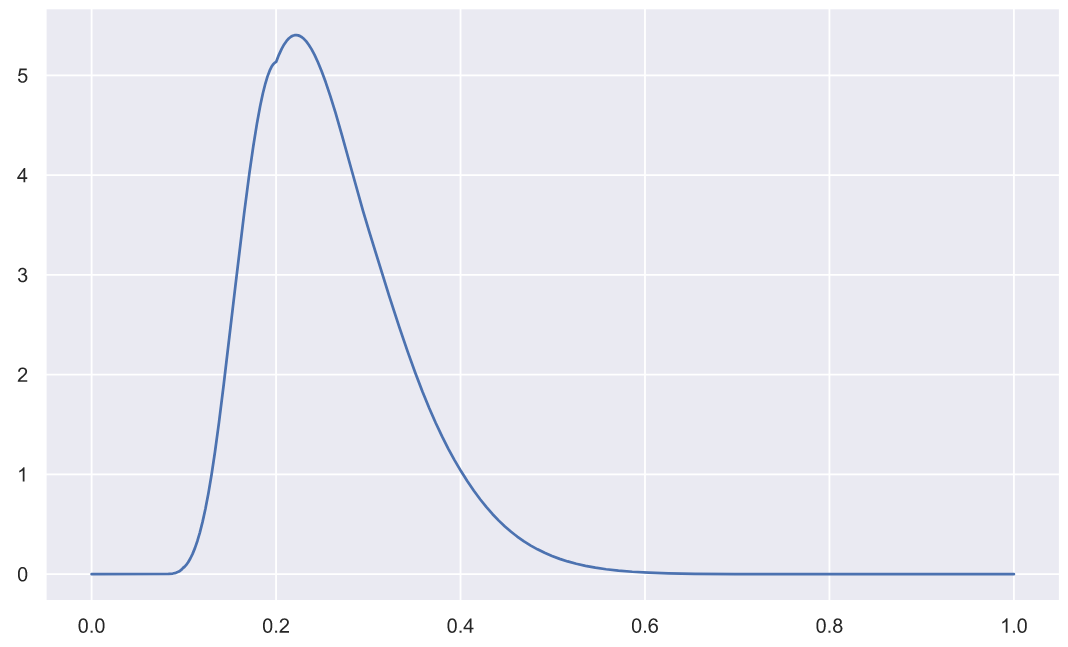
p-value:
pvalue = stats.kstwo.sf(statistic, n) pvalue
0.9971279018404035
, . , , ecdf_le ( ). , ecdf_le . , "" , seaborn, , .
, , -, , : " ?" , : " , ?" , . , - , , ? , . , - ? , .
Los artículos científicos y técnicos no son fáciles de leer, pero escribirlos es aún más tedioso. Me gustaría transmitir algunas ideas complejas de una manera sencilla y relajada. Me gustaría esperar poder hacerlo al menos un poco.
Sin embargo, sea como sea, ¡seguiremos buceando! En la canción de Eminem "Mi nombre es" tengo una frase favorita "Acabo de beber una quinta parte de vodka, ¿me atreves a conducir? (Adelante)", que encaja muy bien para toda la inmersión.
Como de costumbre, presiono F5 y espero sus comentarios.