No sé ustedes, pero las estadísticas no fueron fáciles para mí. Además, fue "dado" - todavía se dice en voz alta. Sí, resultó que puede pasar bastante tiempo en los manuales de capacitación, profundizando de alguna manera en el significado de las fórmulas de cuatro pisos y, a veces, ni siquiera comprender los resultados, pero aún así. Ir y no obtener ningún placer: todo parece estar claro, pero la sensación de que "no estás del todo en el tema" todavía no desaparece. Durante un tiempo traté de leer libros sobre R y no es que fuera completamente infructuoso, pero tampoco "disparar". Encontré el libro más genial "Estadísticas para todos" de Sarah Boslaf, lo leí ... todavía había algunos matices, cuyo significado no se comprende del todo.
En general, como habrás adivinado, este artículo es de la serie "Trato de explicar con los dedos para resolverlo yo mismo". Entonces, si no eres indiferente a las estadísticas, por favor, debajo de cat.
, , . , :
" " . . , . . ;
" " . . . , , " ". , - , " " . , , - - . :
" " . . . . "", . .
" " . , , , . , , , . , . , , . , , . .
, ", , , ", , , "".
(- , ) " - ". NumPy, matplotlib, seaborn SciPy - . , .
... , - :
import numpy as np
from scipy import stats
import matplotlib.pyplot as plt
#import seaborn as sns
from pylab import rcParams
#sns.set()
rcParams['figure.figsize'] = 10, 6
%config InlineBackend.figure_format = 'svg'
np.random.seed(42)
, , , , , . :
norm_rv = stats.norm(loc=30, scale=5)
samples = np.trunc(norm_rv.rvs(365))
samples[:30]
array([32., 29., 33., 37., 28., 28., 37., 33., 27., 32., 27., 27., 31.,
20., 21., 27., 24., 31., 25., 22., 37., 28., 30., 22., 27., 30.,
24., 31., 26., 28.])
:
samples.mean(), samples.std()
(29.52054794520548, 4.77410133275075)
, - .
, , :
sns.histplot(x=samples, discrete=True);

, ,
. , , ? ? , , :
norm_rv = stats.norm(loc=30, scale=5)
beta_rv = stats.beta(a=5, b=5, loc=14, scale=32)
gamma_rv = stats.gamma(a = 20, loc = 7, scale=1.2)
tri_rv = stats.triang(c=0.5, loc=17, scale=26)
x = np.linspace(10, 50, 300)
sns.lineplot(x = x, y = norm_rv.pdf(x), color='r', label='norm')
sns.lineplot(x = x, y = beta_rv.pdf(x), color='g', label='beta')
sns.lineplot(x = x, y = gamma_rv.pdf(x), color='k', label='gamma')
sns.lineplot(x = x, y = tri_rv.pdf(x), color='b', label='triang')
sns.histplot(x=samples, discrete=True, stat='probability',
alpha=0.2);

?
, :
;
- ;
;
;
;
, .
, , ( , - . ?). , , , .. . , , . , :
unif_rv = stats.uniform(loc=0, scale=4)
unif_rv.rvs()
0.8943833540778106
, - -- :
unif_rv.rvs(size=3)
array([3.85289016, 0.0486179 , 3.87951531])
, 15 , . , :
- , - .. ;
, , , - .. ;
, , , , - - .. .
15 : , , ,
:
, -
? 10 :
Y_samples = [unif_rv.rvs(size=15).sum() for i in range(10000)]
sns.histplot(x=Y_samples);

, ? , , : .
, , :
. , ;
- . - ;
. ;
. , ;
. ?;
, . , .
, , . , , :
:
? , . , 365- , , , .
Z-
, . , . , , , 40 , 35 .
? , ? , , , , 27, 31, 35 , 23 38 . , 365 , 20 45 .
, , - 25 35 . , :
N = 5000
t_data = norm_rv.rvs(N)
t_data[(25 < t_data) & (t_data < 35)].size/N
0.7008
- 25 35 . 40 ?
t_data[t_data > 40].size/N
0.0248
40 . 40 . , :
0.02**3
8.000000000000001e-06
, - . Z-.
Z- :
- , .. -
, ,
. , .. , 30 5 . Z- :
? , . Z-? "":
fig, ax = plt.subplots()
x = np.linspace(norm_rv.ppf(0.001), norm_rv.ppf(0.999), 200)
ax.vlines(40.5, 0, 0.1, color='k', lw=2)
sns.lineplot(x=x, y=norm_rv.pdf(x), color='r', lw=3)
sns.histplot(x=samples, stat='probability', discrete=True);

samples, , . . 40 . , . - , .. , , 5000 , , :
np.random.seed(42)
N = 10000
values = np.trunc(norm_rv.rvs(N))
fig, ax = plt.subplots()
v_le_41 = np.histogram(values, np.arange(9.5, 41.5))
v_ge_40 = np.histogram(values, np.arange(40.5, 51.5))
ax.bar(np.arange(10, 41), v_le_41[0]/N, color='0.8')
ax.bar(np.arange(41, 51), v_ge_40[0]/N)
p = np.sum(v_ge_40[0]/N)
ax.set_title('P(Y>40min) = {:.3}'.format(p))
ax.vlines(40.5, 0, 0.08, color='k', lw=1);
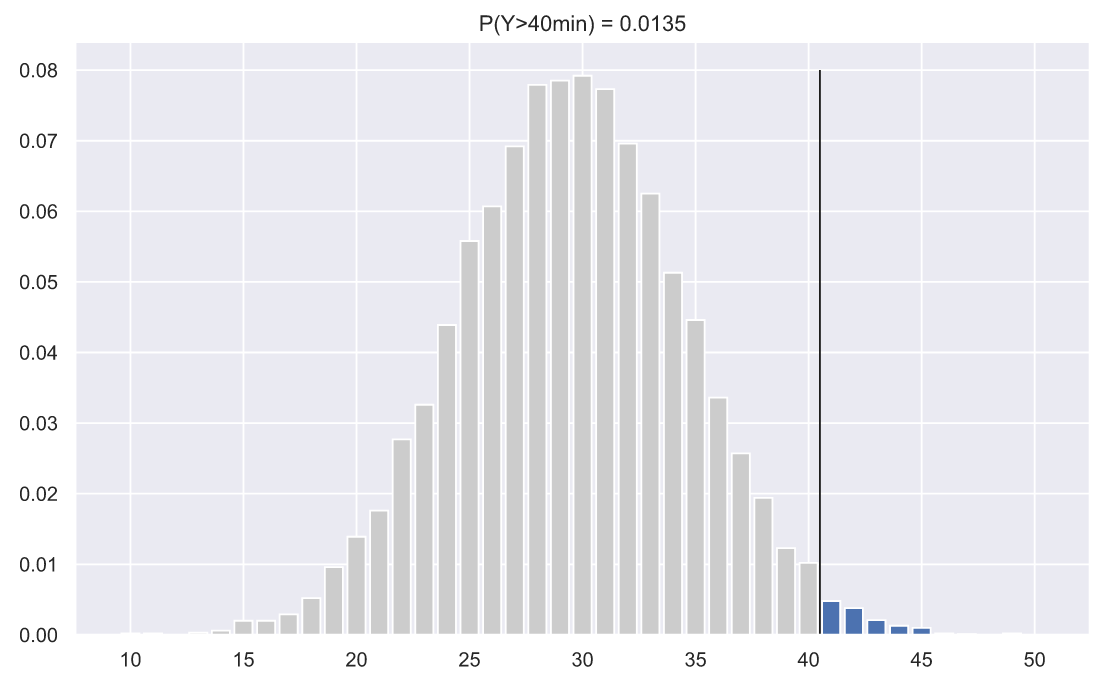
- . , , . , , , . , , 40 :
fig, ax = plt.subplots()
x = np.linspace(norm_rv.ppf(0.0001), norm_rv.ppf(0.9999), 300)
ax.plot(x, norm_rv.pdf(x))
ax.fill_between(x[x>41], norm_rv.pdf(x[x>41]), np.zeros(len(x[x>41])))
p = 1 - norm_rv.cdf(41)
ax.set_title('P(Y>40min) = {:.3}'.format(p))
ax.hlines(0, 10, 50, lw=1, color='k')
ax.vlines(41, 0, 0.08, color='k', lw=1);

. , , - , "", - , . , - , . 40 , .. 41, 42, 43 .. . 41.0. , SciPy , , - . , , - , : , , .. , Z-.
, -
. 99 143 , ? , , :
fig, ax = plt.subplots(nrows=1, ncols=2, figsize = (12, 5))
nrv_hobbit = stats.norm(91, 8)
nrv_gnome = stats.norm(134, 6)
for i, (func, h) in enumerate(zip((nrv_hobbit, nrv_gnome), (99, 143))):
x = np.linspace(func.ppf(0.0001), func.ppf(0.9999), 300)
ax[i].plot(x, func.pdf(x))
ax[i].fill_between(x[x>h], func.pdf(x[x>h]), np.zeros(len(x[x>h])))
p = 1 - func.cdf(h)
ax[i].set_title('P(H > {} ) = {:.3}'.format(h, p))
ax[i].hlines(0, func.ppf(0.0001), func.ppf(0.9999), lw=1, color='k')
ax[i].vlines(h, 0, func.pdf(h), color='k', lw=2)
ax[i].set_ylim(0, 0.07)
fig.suptitle(' , ', fontsize = 20);

, , , ( ), . , . "".
Z-:
fig, ax = plt.subplots()
N_rv = stats.norm()
x = np.linspace(N_rv.ppf(0.0001), N_rv.ppf(0.9999), 300)
ax.plot(x, N_rv.pdf(x))
ax.hlines(0, -4, 4, lw=1, color='k')
ax.vlines(1, 0, 0.4, color='r', lw=2, label='')
ax.vlines(1.5, 0, 0.4, color='g', lw=2, label='')
ax.legend();

Z- , "", .. , Z- . , , , Z- . :
, .
Z- "" , Z- . :
, : , , ; , Z- . Z-, , Z- .
, - - . , . , , Z- - () , . . , , , . - .
Z-
, . Z- 40 :
, , :
fig, ax = plt.subplots()
N_rv = stats.norm()
x = np.linspace(N_rv.ppf(0.0001), N_rv.ppf(0.9999), 300)
ax.plot(x, N_rv.pdf(x))
ax.hlines(0, -4, 4, lw=1, color='k')
ax.vlines(2, 0, 0.4, color='g', lw=2);

, . , , . , - !
, - . 365- , , .. , . . , , , . 5000 :
sns.histplot(np.trunc(norm_rv.rvs(size=(N, 3))).mean(axis=1),
bins=np.arange(19, 42));

, 40 - . :
;
- , .. - , .
Z- (Z = 2), ( ) :
1 - N_rv.cdf(2)
0.02275013194817921
, 40 :
(1 - N_rv.cdf(2))**3
1.1774751750476762e-05
Z-, - Z-:
- ,
,
- .
35 , Z- :
Z-, Z- , . , ,
[25; 35] . :
N = 10000
means = np.trunc(norm_rv.rvs(size=(N, 3))).mean(axis=1)
means[(means>=25)&(means<=35)].size/N
0.9241
N = 10000
fig, ax = plt.subplots()
means = np.trunc(norm_rv.rvs(size=(N, 3))).mean(axis=1)
h = np.histogram(means, np.arange(19, 41))
ax.bar(np.arange(20, 25), h[0][0:5]/N, color='0.5')
ax.bar(np.arange(25, 36), h[0][5:16]/N)
ax.bar(np.arange(36, 41), h[0][16:22]/N, color='0.5')
p = np.sum(h[0][6:16]/N)
ax.set_title('P(25min < Y < 35min) = {:.3}'.format(p))
ax.vlines([24.5 ,35.5], 0, 0.08, color='k', lw=1);

, :
x, n, mu, sigma = 35, 3, 30, 5
z = abs((x - mu)/(sigma/n**0.5))
N_rv = stats.norm()
fig, ax = plt.subplots()
x = np.linspace(N_rv.ppf(0.0001), N_rv.ppf(0.9999), 300)
ax.plot(x, N_rv.pdf(x))
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
ax.vlines([-z, z], 0, 0.4, color='g', lw=2)
x_z = x[(x>-z) & (x<z)] # & (x<z)
ax.fill_between(x_z, N_rv.pdf(x_z), np.zeros(len(x_z)), alpha=0.3)
p = N_rv.cdf(z) - N_rv.cdf(-z)
ax.set_title('P({:.3} < z < {:.3}) = {:.3}'.format(-z, z, p));

, Z- - , -
. 5, 30 100 , , [29; 31]? :
fig, ax = plt.subplots(nrows=1, ncols=3, figsize = (12, 4))
for i, n in enumerate([5, 30, 100]):
x, mu, sigma = 31, 30, 5
z = abs((x - mu)/(sigma/n**0.5))
N_rv = stats.norm()
x = np.linspace(N_rv.ppf(0.0001), N_rv.ppf(0.9999), 300)
ax[i].plot(x, N_rv.pdf(x))
ax[i].hlines(0, x.min(), x.max(), lw=1, color='k')
ax[i].vlines([-z, z], 0, 0.4, color='g', lw=2)
x_z = x[(x>-z) & (x<z)] # & (x<z)
ax[i].fill_between(x_z, N_rv.pdf(x_z), np.zeros(len(x_z)), alpha=0.3)
p = N_rv.cdf(z) - N_rv.cdf(-z)
ax[i].set_title('n = {}, z = {:.3}, p = {:.3}'.format(n, z, p));
fig.suptitle(r'Z- $\bar{x} = 31$', fontsize = 20, y=1.02);

5 [29;31] , . 30 . - 1 .
, : 30 , 31 5, 30 100 ? , n=5 , , , .
,
. ? , 100 31 , , 30 .
, 3 ? ? - . 40 , Z- 3.81, 1:
x, n, mu, sigma = 41, 3, 30, 5
z = abs((x - mu)/(sigma/3**0.5))
N_rv = stats.norm()
fig, ax = plt.subplots()
x = np.linspace(N_rv.ppf(1e-5), N_rv.ppf(1-1e-5), 300)
ax.plot(x, N_rv.pdf(x))
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
ax.vlines([-z, z], 0, 0.4, color='g', lw=2)
x_z = x[(x>-z) & (x<z)] # & (x<z)
ax.fill_between(x_z, N_rv.pdf(x_z), np.zeros(len(x_z)), alpha=0.3)
p = N_rv.cdf(z) - N_rv.cdf(-z)
ax.set_title('P({:.3} < z < {:.3}) = {:.3}'.format(-z, z, p));

, , ,
10 . :
"" ;
.
? .
p-value
Z- ,
, . ,
. Z-, . ,
[29;31] 0.35. 1−0.35=0.65. ,
, - .
, 0.65, - p-value , Z-, :
x, n, mu, sigma = 31, 5, 30, 5
z = abs((x - mu)/(sigma/n**0.5))
N_rv = stats.norm()
fig, ax = plt.subplots()
x = np.linspace(N_rv.ppf(0.0001), N_rv.ppf(0.9999), 300)
ax.plot(x, N_rv.pdf(x))
ax.hlines(0, x.min(), x.max(), lw=1, color='k')
ax.vlines([-z, z], 0, 0.4, color='g', lw=2)
x_le_z, x_ge_z = x[x<-z], x[x>z]
ax.fill_between(x_le_z, N_rv.pdf(x_le_z), np.zeros(len(x_le_z)), alpha=0.3, color='b')
ax.fill_between(x_ge_z, N_rv.pdf(x_ge_z), np.zeros(len(x_ge_z)), alpha=0.3, color='b')
p = N_rv.cdf(z) - N_rv.cdf(-z)
ax.set_title('P({:.3} < z < {:.3}) = {:.3}'.format(-z, z, p));

p-value , . p-value , .. . - , p-value , . , , , 0.05, , p-value . , ,
. ,
, , 5 , ..
:
1 - (N_rv.cdf(5) - N_rv.cdf(-5))
5.733031438470704e-07
, , . , , , . , , - "" . , ( ) . , . , , , .
, , . , , , 31 , 30 , 100 .
, , Z- : , , , .
No parece muy plausible, pero echemos un vistazo. Generaremos 1000 valores a partir de las distribuciones uniforme, exponencial y de Laplace, y luego, secuencialmente, para cada distribución, construiremos gráficos kde de las distribuciones del valor medio de muestras de diferentes tamaños:
Código GIF
import matplotlib.animation as animation
fig, axes = plt.subplots(nrows=2, ncols=3, figsize = (12, 7))
unif_rv = stats.uniform(loc=10, scale=10)
exp_rv = stats.expon(loc=10, scale=1.5)
lapl_rv = stats.laplace(loc=15)
np.random.seed(42)
unif_data = unif_rv.rvs(size=1000)
exp_data = exp_rv.rvs(size = 1000)
lapl_data = lapl_rv.rvs(size=1000)
title = ['Uniform', 'Exponential', 'Laplace']
data = [unif_data, exp_data, lapl_data]
y_max = [60, 310, 310]
n = [3, 10, 30, 50]
for i, ax in enumerate(axes[0]):
sns.histplot(data[i], bins=20, alpha=0.4, ax=ax)
ax.vlines(data[i].mean(), 0, y_max[i], color='r')
ax.set_xlim(10, 20)
ax.set_title(title[i])
def animate(i):
for ax in axes[1]:
ax.clear()
for j in range(3):
rand_idx = np.random.randint(0, 1000, size=(1000, n[i]))
means = data[j][rand_idx].mean(axis=1)
sns.kdeplot(means, ax=axes[1][j])
axes[1][j].vlines(means.mean(), 0, 2, color='r')
axes[1][j].set_xlim(10, 20)
axes[1][j].set_ylim(0, 2.1)
axes[1][j].set_title('n = ' + str(n[i]))
fig.set_figwidth(15)
fig.set_figheight(8)
return axes[1][0], axes[1][1],axes[1][2]
dist_animation = animation.FuncAnimation(fig,
animate,
frames=np.arange(4),
interval = 200,
repeat = False)
# gif :
dist_animation.save('dist_means.gif',
writer='imagemagick',
fps=1)
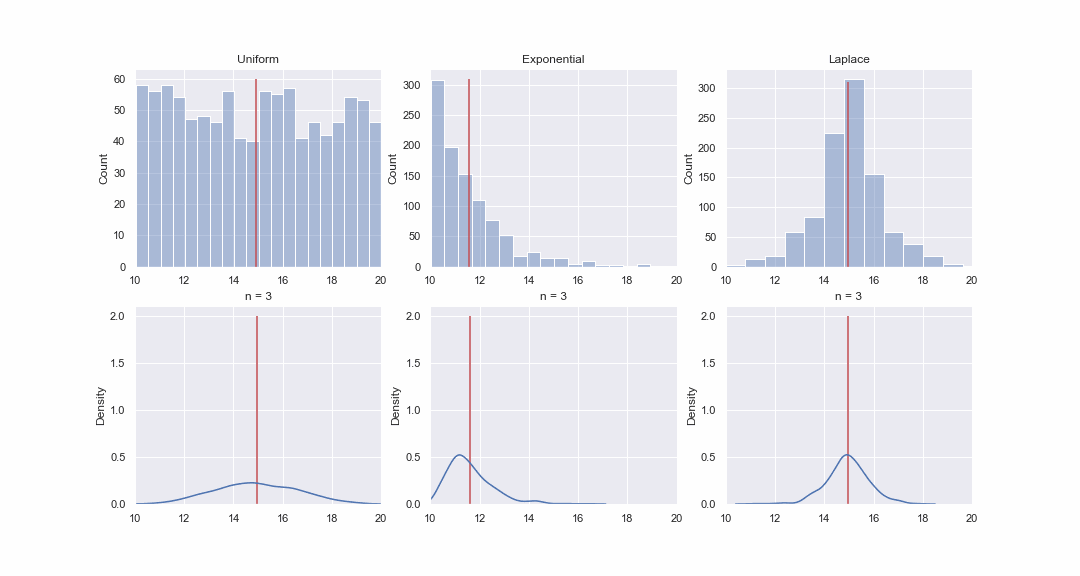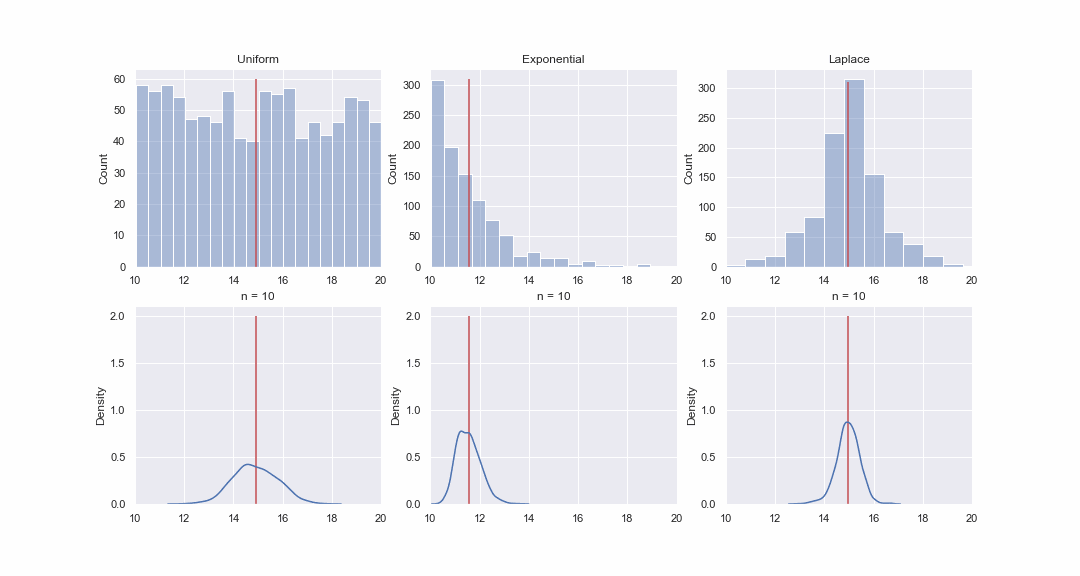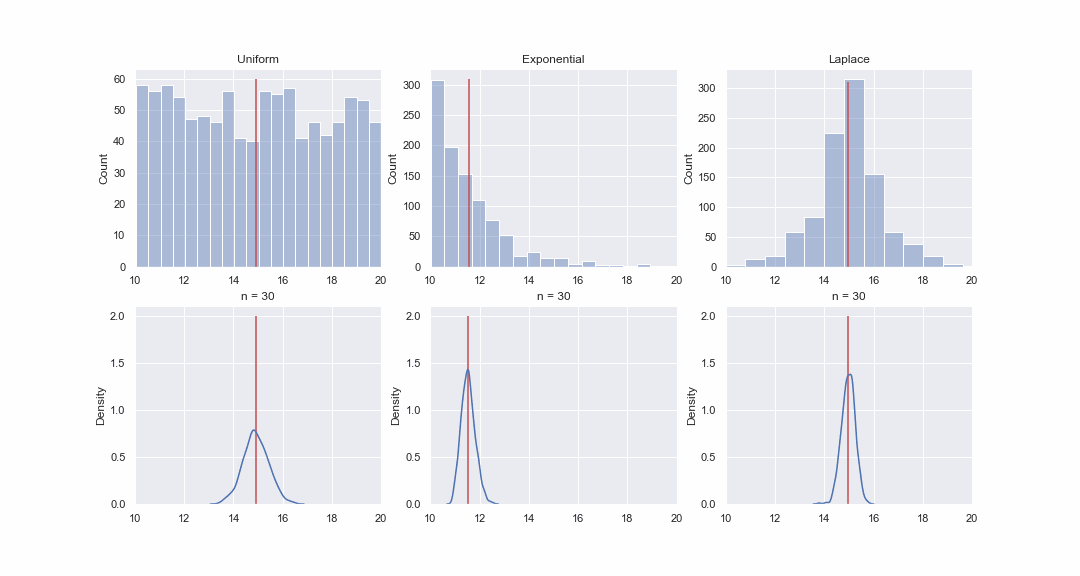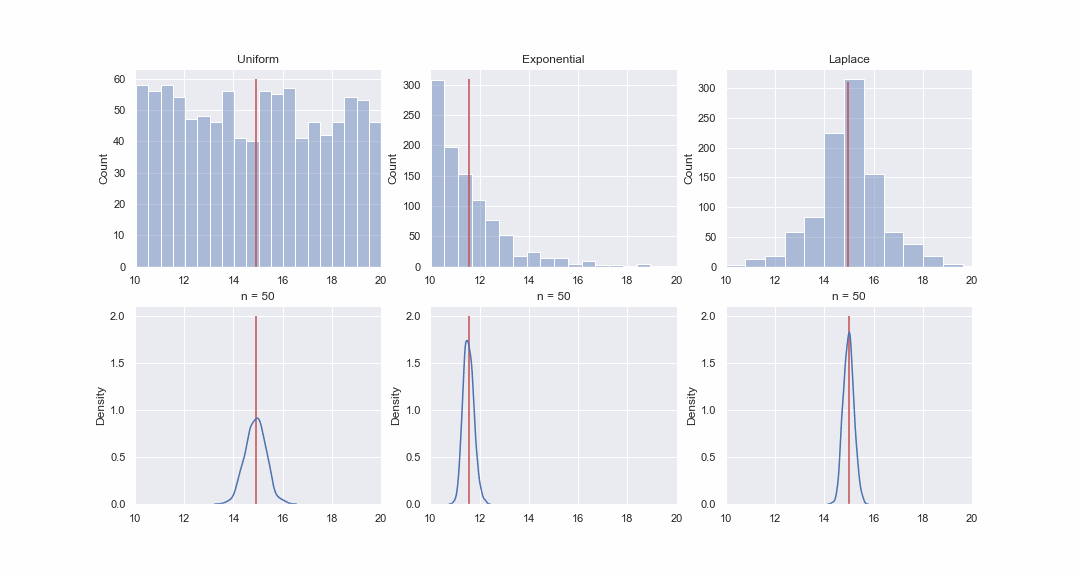
Por supuesto, la imagen no es una prueba en absoluto, pero si lo desea, puede ejecutar las pruebas de normalidad usted mismo. Sin embargo, en el próximo artículo solo haremos pruebas y probaremos hipótesis.
En general, gracias por su atención. Presiono F5 y espero sus comentarios y los suyos.