Para la genética del trigo, una tarea importante es determinar la ploidía (el número de conjuntos idénticos de cromosomas en el núcleo celular). El enfoque clásico para resolver este problema se basa en el uso de métodos genéticos moleculares, que son costosos y laboriosos. La determinación de los tipos de plantas solo es posible en condiciones de laboratorio. Por lo tanto, en este trabajo, probamos la hipótesis: ¿es posible determinar la ploidía del trigo utilizando métodos de visión por computadora, solo sobre la base de una imagen de una oreja?

Descripción de datos
Para solucionar el problema, incluso antes del inicio del taller, se preparó un conjunto de datos en el que se conocía la ploidía de cada especie vegetal. En total, teníamos a nuestra disposición 2344 fotografías de hexaploides y 1259 tetraproides.
La mayoría de las plantas se fotografiaron utilizando dos protocolos. El primer caso, en una mesa en una proyección, el segundo, en una pinza para ropa en 4 proyecciones. En las fotografías, la paleta de colores del comprobador de color siempre estuvo presente , es necesaria para normalizar los colores y determinar la escala.

Un total de 3603 fotos con 644 números de semillas únicos. El conjunto de datos contiene 20 especies de trigo: 10 hexaploides, 10 tetraploides; 496 genotipos únicos; 10 vegetación única. Las plantas se cultivaron entre 2015 y 2018 en invernaderos.ICG SB RAS . El material biológico fue proporcionado por el académico Nikolai Petrovich Goncharov .
Validación
Una planta en nuestro conjunto de datos puede corresponder a hasta 5 fotografías tomadas usando diferentes protocolos y en diferentes proyecciones. Dividimos los datos en 3 conjuntos estratificados: tren (muestra de entrenamiento), válido (muestra de validación) y espera (muestra retrasada), en proporciones del 60%, 20% y 20%, respectivamente. Al dividir, se tuvo en cuenta que todas las fotografías de un determinado genotipo aparecerían siempre en una submuestra. Este esquema de validación se utilizó para todos los modelos entrenados.
Probar métodos clásicos de CV y ML
El primer enfoque que usamos para resolver el problema se basa en el algoritmo existente que desarrollamos anteriormente. El algoritmo permite extraer un conjunto fijo de diferentes características cuantitativas de cada imagen. Por ejemplo, la longitud de la oreja, el área de las aristas , etc. Para obtener una descripción detallada del algoritmo, consulte Genaev et al., Morphometry of the Wheat Spike by Analyzing 2D Images, 2019 . Utilizando este algoritmo y métodos de aprendizaje automático, entrenamos varios modelos para predecir los tipos de ploidía.
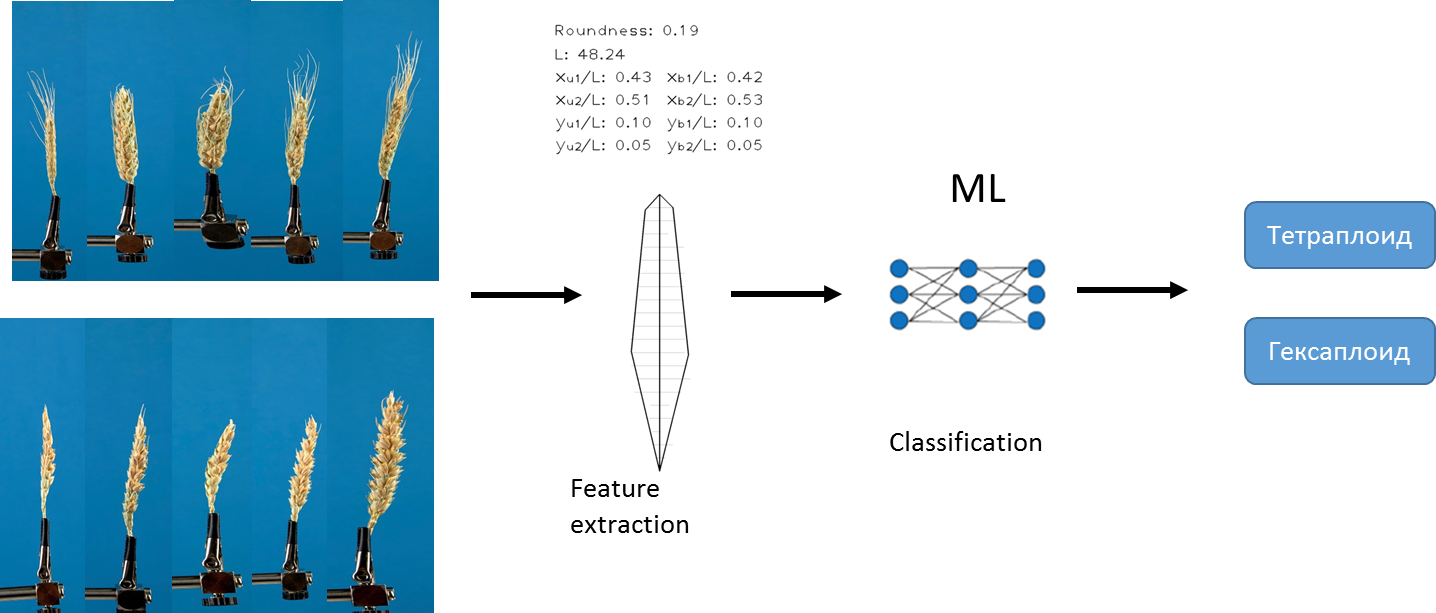
Usamos métodos de regresión logística , bosque aleatorio y aumento de gradiente . Los datos se han normalizado previamente... Elegimos el AUC como medida de precisión .
| Método | Entrenar | Válido | Holdout |
| Regresión logística | 0,77 | 0,70 | 0,72 |
| Bosque aleatorio | 1,00 | 0,83 | 0,82 |
| Impulsar | 0,99 | 0,83 | 0,85 |
La mejor precisión en el muestreo diferido se demostró mediante el método de aumento de gradiente; utilizamos la implementación de CatBoost.
Interpretando los resultados
Para cada modelo, recibimos una estimación de la "importancia" de cada rasgo. Como resultado, obtuvimos una lista de todas nuestras características, clasificadas por significancia y seleccionamos las 10 características principales: área de aristas, índice de circularidad, redondez, perímetro, longitud del tallo, xu2, L, xb2, yu2, ybm. (se puede encontrar una descripción de cada característica aquí ).
La longitud y el perímetro de la oreja son ejemplos de características importantes. Las distribuciones de los valores de estos rasgos en tetraploides y hexaploides se muestran en los histogramas. Se puede ver que la distribución de los hexaploides se desplaza hacia valores más altos.

Hemos agrupado las 10 características principales utilizando el método t-SNE
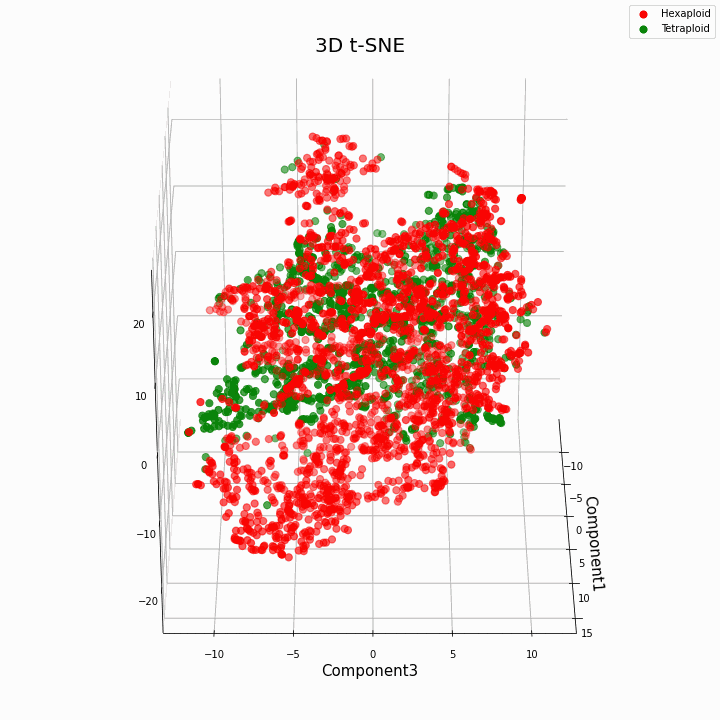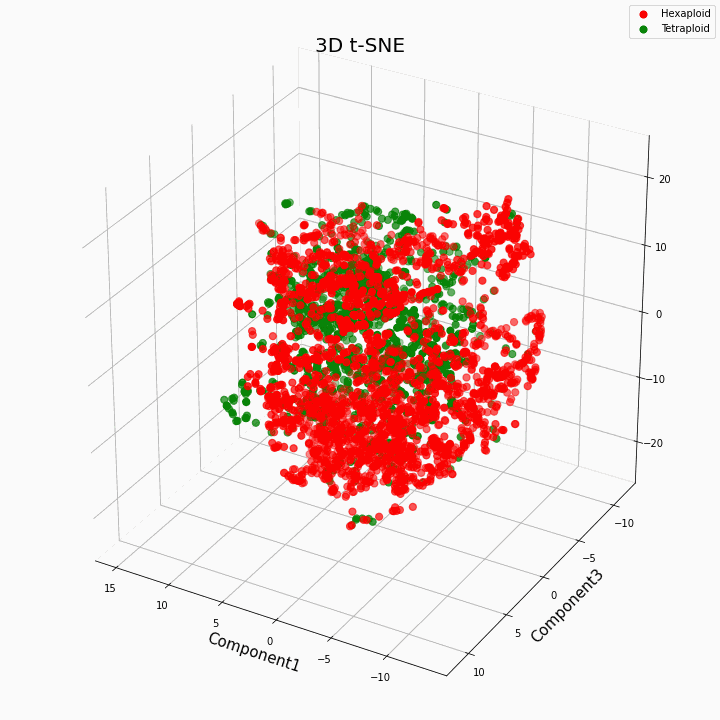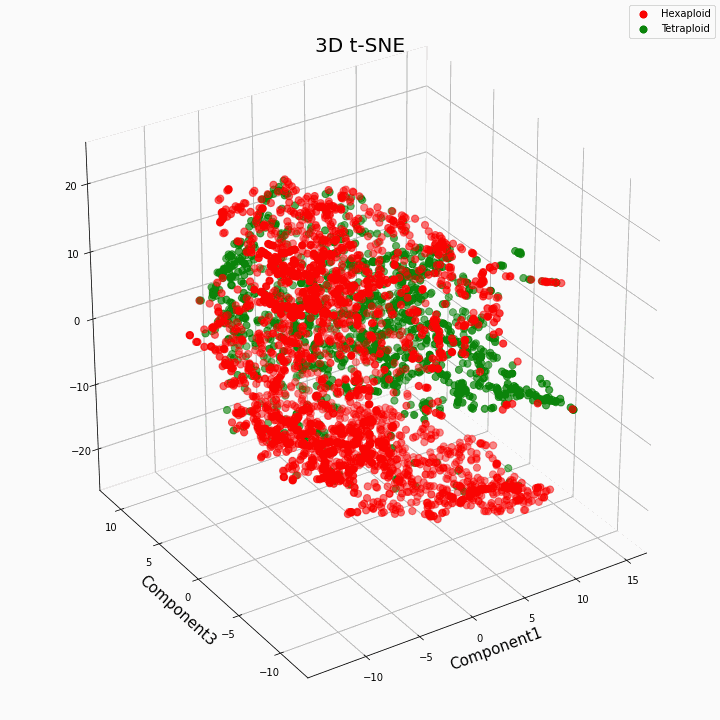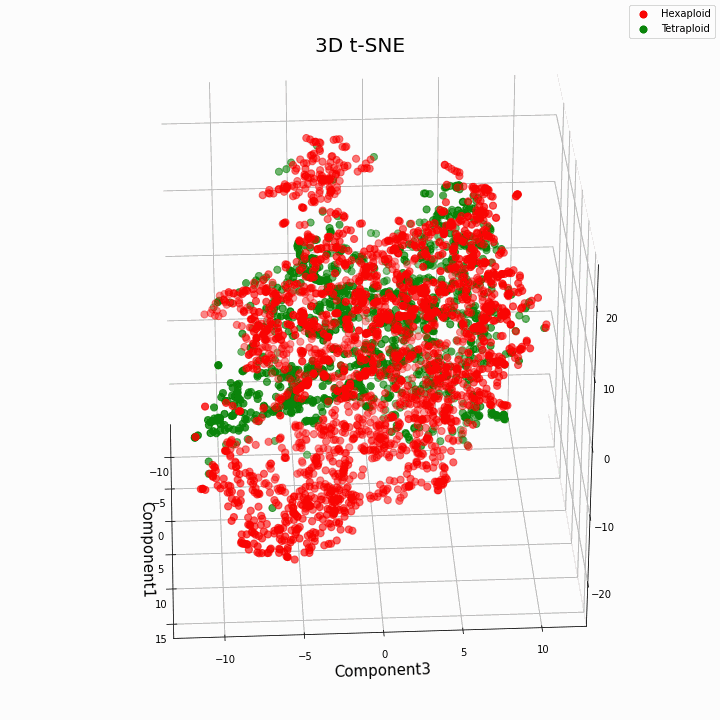
En general, una mayor ploidía da valores más variables de rasgos. Los hexaploides se caracterizan por una mayor dispersión / varianza de los valores del rasgo. Esto se debe a que el número de copias de genes en hexaploides es mayor y, por tanto, aumenta el número de variantes del "trabajo" de estos genes.
Para confirmar nuestra hipótesis de mayor variabilidad fenotípica en hexaploides, aplicamos el estadístico F. El estadístico F da la importancia de las diferencias en las varianzas de las dos distribuciones. Consideramos los casos en los que el valor p es menor que 0.05 para refutar la hipótesis nula de que no existen diferencias entre las dos distribuciones. Realizamos esta prueba de forma independiente para cada rasgo. Condiciones de prueba: debe haber una muestra de observaciones independientes (en el caso de varias imágenes, este no es el caso) y distribuciones normales. Para cumplir estas condiciones, probamos una imagen de cada oído. Tomaron fotografías en una sola proyección según el protocolo “sobre la mesa”. Los resultados se muestran en la tabla. Se puede observar que la varianza para hexaploides y tetraploides tiene diferencias significativas para 7 caracteres. Además, en todos los casos, el valor de la dispersión es mayor en hexaploides.La mayor variabilidad fenotípica de los hexaploides puede explicarse por el gran número de copias de un gen.
| Name | F-statistic | p-value | Disp Hexaploid | Disp Tetraploid |
| Awns area | 0.376 | 1.000 | 1.415 | 3.763 |
| Circularity index | 1.188 | 0.065 | 0.959 | 0.807 |
| Roundness | 1.828 | 0.000 | 1.312 | 0.718 |
| Perimeter | 1.570 | 0.000 | 1.080 | 0.688 |
| Stem length | 3.500 | 0.000 | 1.320 | 0.377 |
| xu2 | 3.928 | 0.000 | 1.336 | 0.340 |
| L | 3.500 | 0.000 | 1.320 | 0.377 |
| xb2 | 4.437 | 0.000 | 1.331 | 0.300 |
| yu2 | 4.275 | 0.000 | 2.491 | 0.583 |
| ybm | 1.081 | 0.248 | 0.695 | 0.643 |
Nuestros datos incluyen 20 especies de plantas. 10 trigo hexaploide y 10 tetraploide.
Hemos coloreado los resultados de la agrupación para que el color + la forma de cada punto corresponda a una vista específica.

La mayoría de las especies ocupan áreas bastante compactas en el gráfico. Aunque estas áreas pueden superponerse mucho con otras. Por otro lado, dentro de una especie puede haber agrupaciones claramente definidas, por ejemplo, para T compactum, T petropavlovskyi.
Hicimos un promedio de los valores de cada especie para 10 características, obteniendo una tabla de 20 por 10. Donde cada una de las 20 especies corresponde a un vector de 10 características. Para estos datos, se construyó una matriz de correlación y se realizó un análisis de conglomerados jerárquicos. Los cuadrados azules del gráfico corresponden a tetraploides.

En el árbol construido, en general, las especies de trigo se dividieron en tetraploides y hexaploides. Las especies hexaploides se dividen claramente en dos grupos: de pelo medio - T. macha, T. aestivum, T. yunnanense y de pelo largo - T. vavilovii, T. petropavlovskyi, T. spelta. La única excepción es que la única especie silvestre poliploide (tetraploide) T. dicoccoides se clasificó como hexaploide.
Al mismo tiempo, las especies tetraploides incluían el trigo hexaploide con un tipo de mazorca compacta: T. compactum, T. antiquorum y T. sphaerococcum, y la línea isogénica artificial ANK-23 del trigo blando.
Probar CNN

Para resolver el problema de determinar la ploidía del trigo a partir de la imagen de una oreja, entrenamos una red neuronal convolucional de la arquitectura EfficientNet B0 con pesos preentrenados en ImageNet. CrossEntropyLoss se utilizó como función de pérdida; Optimizador de Adam; el tamaño de un lote es 16; las imágenes se redimensionaron a 224x224; la tasa de aprendizaje se cambió de acuerdo con la estrategia fit_one_cycle con un lr inicial = 1e-4. Entrenamos la red durante 10 épocas, aplicando los siguientes aumentos al azar: rotaciones de -20 +20 grados, cambio de brillo, contraste, saturación, reflejo. El mejor modelo se eligió según la métrica AUC , cuyo valor se calculó al final de cada época.
Como resultado, la precisión de la muestra diferida AUC = 0,995 , que corresponde a la puntuación de precisión= 0,987 y un error del 1,3%. Que es un muy buen resultado.
Conclusión
Este trabajo es un buen ejemplo de cómo un equipo de 5 estudiantes y 2 comisarios pueden solucionar un problema biológico urgente y obtener nuevos resultados científicos en pocas semanas.
Me gustaría expresar mi gratitud a todos los participantes de nuestro proyecto: Nikita Prokhoshin , Alexei Prikhodko , Evgeny Zavarzin , Artem Pronozin , Anna Paulish , Evgeny Komyshev, Mikhail Genaev .
Koval Vasily Sergeevich y Kruchinina Yulia Vladimirovna por fotografiar mazorcas de maíz.
Nikolai Petrovich Goncharov y Afonnikov Dmitry Arkadyevich por el material biológico proporcionado y su ayuda en la interpretación de los resultados.
Al Centro de Matemáticas de la Universidad Estatal de Novosibirsk y al Instituto de Citología y Genética del SB RAS por la organización del evento y la potencia informática.

PD: Planeamos preparar la segunda parte del artículo, donde hablaremos sobre la segmentación de una oreja y la selección de espiguillas individuales.